Utiliser la transformation des données DICOM dans les solutions de données de santé
La fonctionnalité de transformation des données DICOM dans les solutions de données de santé vous permet d’ingérer, de stocker et d’analyser les données DICOM (Digital Imaging and Communications in Medicine) de diverses sources. Pour en savoir plus sur la fonctionnalité et comprendre comment la déployer et la configurer, consultez :
- Vue d’ensemble de transformation des données DICOM
- Transformation des métadonnées DICOM mappage
- Déployer et configurer la transformation des données DICOM
La transformation des données DICOM est une fonctionnalité facultative avec les solutions de données de santé dans Microsoft Fabric.
Conditions préalables
Avant d’exécuter le pipeline de transformation des données DICOM, assurez-vous d’avoir effectué les conditions préalables, le processus de déploiement et les étapes de configuration expliqués dans Déployer et configurer la transformation des données DICOM.
Options d’ingestion des données
Cet article fournit des conseils étape par étape sur la façon d’utiliser la capacité de transformation des données DICOM pour ingérer, transformer et unifier l’imagerie DICOM jeu de données. La fonctionnalité prend en charge les deux options d’ingestion suivantes :
Option 1 : Ingestion de bout en bout des fichiers DICOM. Les fichiers DICOM, aux formats natifs (DCM) ou compressés (ZIP), sont ingérés dans la lakehouse. Cette option est appelée option Ingérer.
Option 2 : Intégration avec le service DICOM. L’ingestion est facilitée grâce à l’intégration native avec le service DICOM dans les services de données de santé Azure. Dans cette option, les fichiers DCM sont d’abord transférés du service DICOM des services de données de santé Azure vers Data Lake Storage Gen2. Le pipeline suit ensuite le modèle d’ingestion Bring Your Own Storage (BYOS) . Cette option est appelée option Services de données de santé Azure (AHDS) .
Pour comprendre les détails de la transformation mappage, consultez Transformation des métadonnées DICOM mappage dans les solutions de données de santé.
Option 1 : Ingestion de bout en bout des fichiers DICOM
Dans cette option, nous ingérons et transformons les données d’imagerie des fichiers DICOM dans les lacs de solutions de données de santé à l’aide du pipeline de données prédéfini. La transformation de bout en bout comprend les étapes consécutives suivantes :
- Ingérer les fichiers DICOM dans OneLake
- Organiser les fichiers DICOM dans OneLake
- Extraire les métadonnées DICOM dans la lakehouse bronze
- Convertir les métadonnées DICOM au format FHIR (Fast Health Interoperability Resources)
- Ingérer les données dans la table delta ImagingStudy de la lakehouse bronze
- Aplatir et transformer les données dans la table delta ImagingStudy de la lakehouse argent
- Convertir et ingérer les données dans la table Image_Occurrence de la lakehouse or (facultatif)
Astuce
Cette option d’ingestion utilise l’exemple 340ImagingStudies jeu de données qui contient des fichiers ZIP compressés. Vous pouvez également ingérer des fichiers DICOM directement dans leur format DCM natif en les plaçant dans le dossier Ingest . Dans les fichiers ZIP, les fichiers DCM peuvent être structurés en plusieurs sous-dossiers imbriqués. Il n’y a aucune limite au nombre de fichiers DCM ou au nombre, à la profondeur et à l’imbrication des sous-dossiers dans les fichiers ZIP ingérés. Pour plus d’informations sur les limites de taille de fichier, voir Taille du fichier d’ingestion.
étape 1 : Ingérer des fichiers DICOM dans OneLake
Le dossier Ingérer dans la lakehouse bronze représente un dossier de dépôt (file d’attente). Vous pouvez déposer les fichiers DICOM dans ce dossier. Les fichiers sont ensuite déplacés vers une structure de dossiers organisée au sein de la lakehouse bronze.
Accédez au dossier
Ingest\Imaging\DICOM\DICOM-HDSdans le bronze lakehouse.Sélectionner ... (ellipse) >Télécharger>Télécharger le dossier.
Sélectionner et téléchargez les 340ImagingStudies images jeu de données depuis le dossier SampleData dans
SampleData\Imaging\DICOM\DICOM-HDS. Vous pouvez également utiliser l’explorateur de fichiers OneLake ou l’explorateur de stockage Azure pour télécharger l’exemple jeu de données.
étape 2 : Exécuter le pipeline de données d’imagerie
Après avoir déplacé les fichiers DCM/ZIP vers le dossier Ingest dans le fichier bronze lakehouse, vous pouvez maintenant exécuter le pipeline de données d’imagerie pour organiser et traiter les données dans le fichier argent lakehouse.
Dans vos solutions de données de santé environnement, ouvrez le pipeline de données healthcare#_msft_imaging_with_clinical_foundation_ingestion .
Sélectionner appuyez sur le bouton Exécuter pour commencer le traitement des données d’imagerie du bronze à l’argent lakehouse.
Ce pipeline de données exécute séquentiellement cinq blocs-notes : trois déployés dans le cadre de la capacité des fondations de données de santé et deux à partir de la capacité de transformation des données DICOM. Pour en savoir plus sur ces blocs-notes, consultez Transformation des données DICOM : artefacts.
étape 3 : Exécutez le carnet de transformation de l’argent en or
Note
Cette transformation étape est facultative. Utilisez-le uniquement si vous devez transformer davantage vos données DICOM au format de modèle de données commun (CDM) du Partenariat pour les résultats médicaux observationnels (OMOP). Sinon, vous pouvez ignorer cette étape.
Avant d’exécuter cette transformation, déployez et configurez la OMOP capacité de transformations dans les solutions de données de santé.
Une fois le pipeline d’imagerie exécuté, vos données d’imagerie se transforment en argent lakehouse. Le lakehouse argenté sert de pointer initial où les données de diverses modalités commencent à converger de manière structurée. Pour transformer davantage vos données en OMOP norme de recherche à utiliser dans la capacité Découvrir et créer des cohortes (version préliminaire) , exécutez le bloc-notes de transformation de l’argent à l’or.
Dans vos solutions de données de santé environnement, ouvrez le bloc-notes healthcare#_msft_omop_silver_gold_transformation .
Ce bloc-notes utilise les API des solutions de données de santé pour transformer les ressources de l’argent lakehouse en tables delta CDM dans l’or lakehouse. OMOP OMOP Par défaut, il n’est pas nécessaire d’apporter des modifications à la configuration du notebook.
Sélectionnez Exécuter tout pour exécuter le notebook.
Le bloc-notes implémente l’approche de suivi pour suivre et traiter les enregistrements nouveaux ou mis à jour dans la table delta d’ImagingStudy dans le fichier silver lakehouse. OMOP Il transforme les données des tables delta FHIR dans le lakehouse argent (y compris la table ImagingStudy ) en tables delta OMOP correspondantes dans le lakehouse or. (y compris la table Image_Occurrence ). Pour plus d’informations sur cette transformation, voir Transformation mappage pour le tableau delta argent vers or.
Pour des informations détaillées OMOP mappage, consultez FHIR vers OMOP mappage.
étape 4 : Valider les données
Dans les scénarios du monde réel, l’ingestion de données implique des sources avec des niveaux de qualité variables. Le moteur de validation, détaillé dans Validation des données, déclenche intentionnellement des validations sur certaines des données d’échantillon d’imagerie fournies. Les fichiers qui ne sont pas conformes aux normes DICOM sont déplacés vers le dossier Échec et ne sont pas traités. Cependant, une seule défaillance de fichier ne perturbe pas l’ensemble du pipeline, comme le démontrent les données d’échantillon d’imagerie. Le pipeline et les blocs-notes associés s’exécutent correctement, mais le dossier Failed sous Imaging\DICOM\DICOM-HDS\YYYY\MM\DD contient un fichier non conforme. Tous les autres fichiers valides sont traités avec succès, ce qui donne un statut de pipeline globalement réussi. Nous incluons intentionnellement ce fichier non valide dans les données d’échantillon d’imagerie pour illustrer comment le pipeline d’imagerie gère les fichiers non valides et vous aide à identifier les problèmes jeu de données.
Pour confirmer que le pipeline a extrait avec succès toutes les métadonnées des fichiers DICOM bruts, ouvrez le bronze lakehouse, passez à l’analyse SQL point de terminaison et à la requête Sélectionner New SQL.
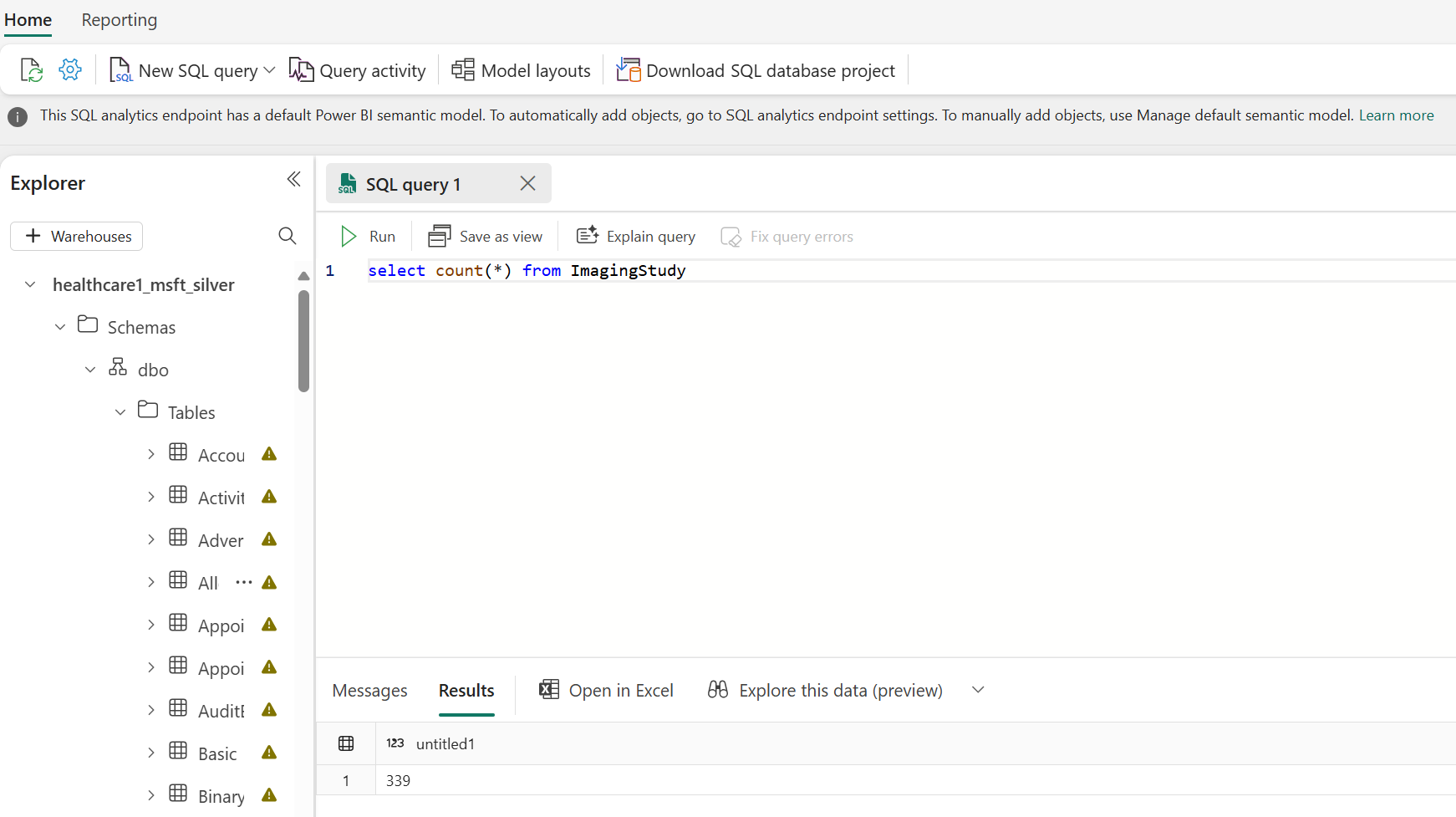
Si le pipeline a fonctionné correctement, vous devez voir 7739 instances DICOM traitées avec succès dans la table ImagingDicom . Pour vérifier, exécutez la requête SQL suivante. Pour un traitement réussi, vous devriez voir 7739 dans le volet Résultats . Ce nombre représente le nombre total d’instances DICOM dans les données d’échantillon, comprenant des données provenant de différentes modalités telles que la tomodensitométrie (TDM) et l’imagerie par résonance magnétique (IRM).
select count(*) from ImagingDicom
Pour confirmer que le pipeline a correctement hydraté les maisons du lac, ouvrez l’argent lakehouse, passez à l’analyse SQL point de terminaison et à la requête Sélectionner New SQL. Pour une exécution correcte du pipeline, vous devez voir 339 ressources ImagingStudy traitées avec succès. Pour vérifier, exécutez la requête SQL suivante. Au départ, nous commençons avec 340 ImagingStudy ressources, mais on rencontre une erreur lors du traitement.
select count(*) from ImagingStudy
Option 2 : Intégration avec le service DICOM
Important
Utilisez cette option de transformation uniquement si vous utilisez le service DICOM Health Data Services Azure et avez déployé l’API DICOM.
Cette approche de transformation étend le modèle BYOS (Bring Your Own Storage) avec le service DICOM Health Data Services. ... Le service DICOM est un sous-ensemble d’ API DICOMweb qui vous permettent de stocker, d’examiner, de rechercher et de supprimer des objets DICOM. Il s’intègre au compte Gen2 lié à votre espace de travail Fabric, afin que le pipeline de transformation puisse accéder directement à vos données DICOM. Azure Data Lake Storage
Alternativement, vous pouvez ignorer l’utilisation de l’API DICOM Azure et ingérer les fichiers DICOM stockés dans votre compte Azure Storage Gen2 (dans ce cas, commencez à partir de étape 5).
Vérifiez et complétez la configuration dans Déployer l’API DICOM dans Azure Health Data Services.
Après avoir déployé le service DICOM Azure, utilisez l’API Store (STOW-RS) pour ingérer les fichiers DCM. Testez-le en téléchargeant un fichier DCM à partir des données d’échantillon d’imagerie à l’aide de l’ explorateur de fichiers OneLake ou de l’ explorateur de stockage Azure.
Selon votre langue préférée, téléchargez les fichiers DCM sur le serveur en utilisant l’une des options suivantes :
Vérifiez si le téléchargement du fichier a réussi :
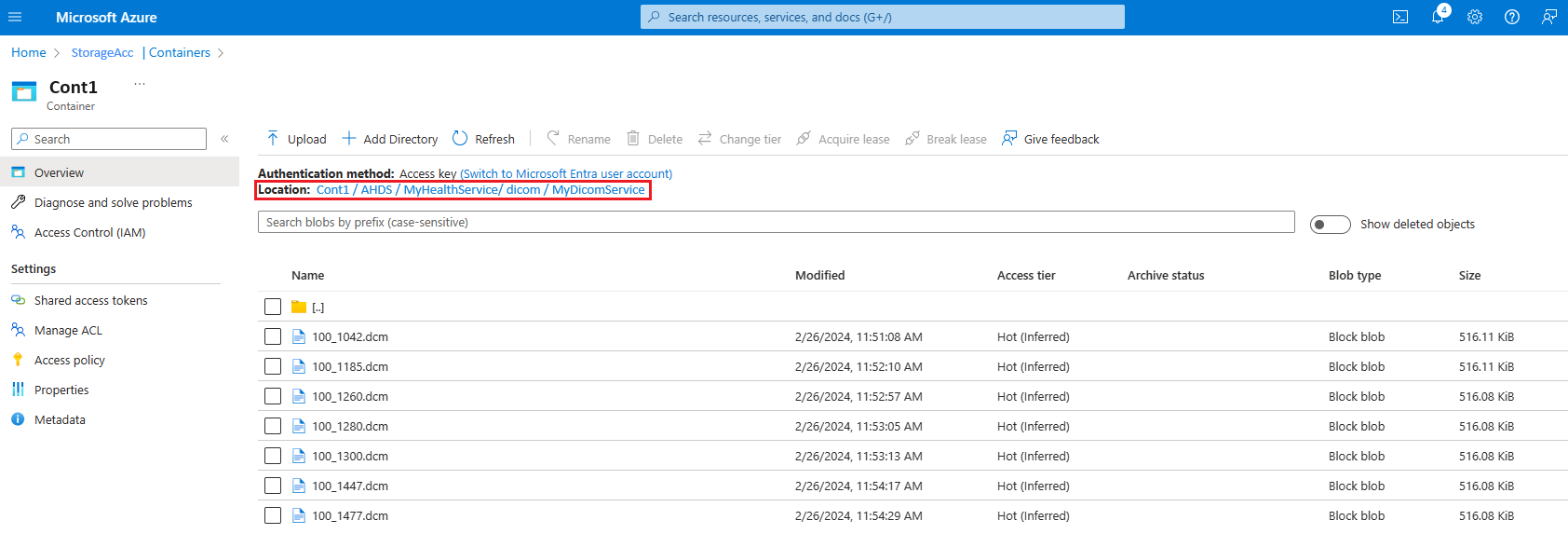
- Dans le portail Azure, Sélectionner le compte de stockage lié au service DICOM.
- Accédez à Conteneurs et suivre le chemin
[ContainerName]/AHDS/[AzureHealthDataServicesWorkspaceName]/dicom/[DICOMServiceName]. - Vérifiez si vous pouvez voir le fichier DCM téléchargé ici.
Note
- Le nom du fichier peut changer lors du téléchargement sur le serveur. Cependant, le contenu du fichier reste inchangé.
- Pour plus d’informations sur les limites de taille de fichier, voir Taille du fichier d’ingestion.
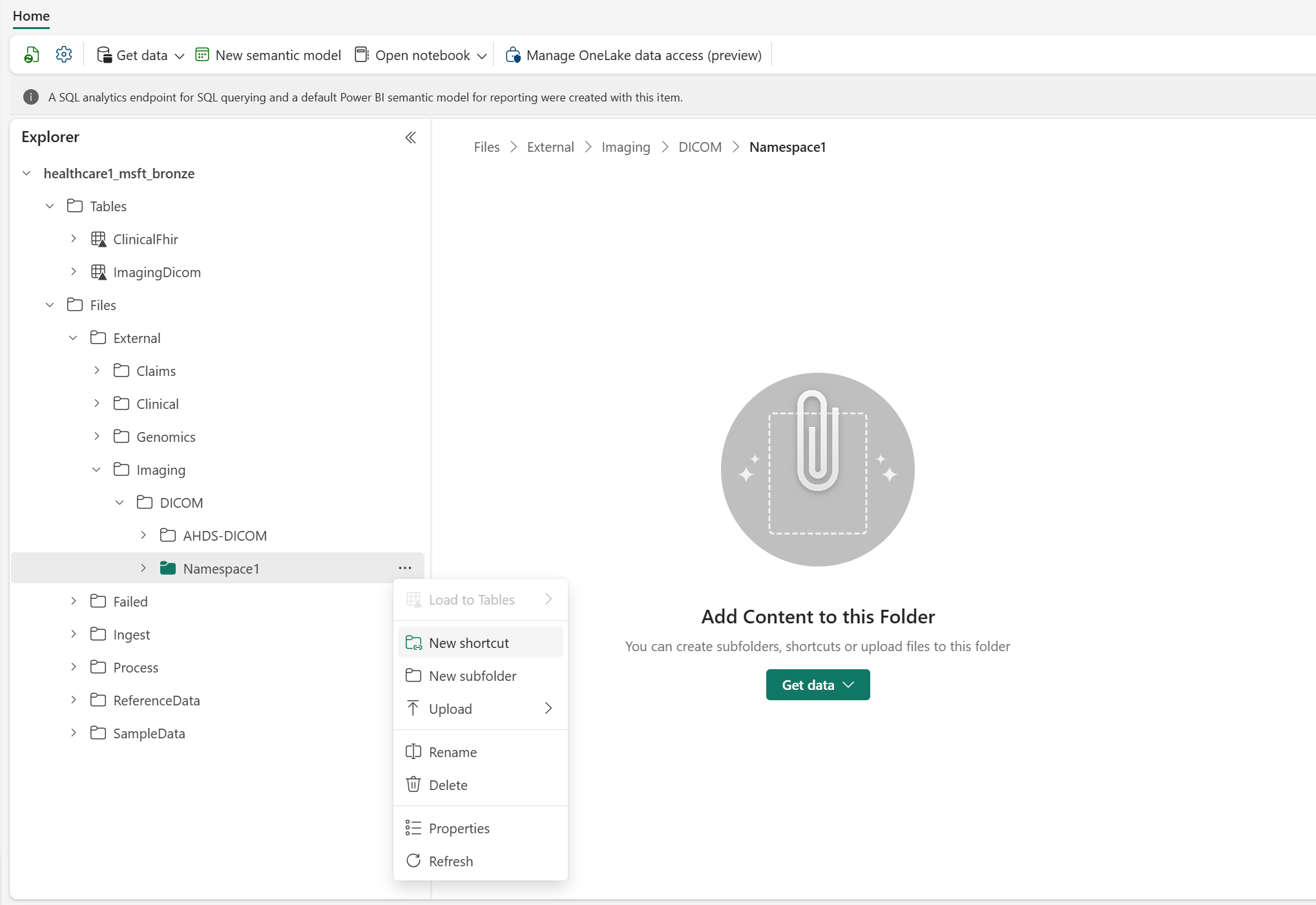
Créez un raccourci dans le bronze lakehouse pour le fichier DICOM stocké dans l’emplacement de stockage Gen2 Data Lake. Suivre les étapes de Créer un Azure Data Lake Storage raccourci Gen2.
- Pour le service DICOM Azure, assurez-vous d’utiliser le compte Data Lake Storage Gen2 créé avec le service.
- Si vous n’utilisez pas le service DICOM Azure, vous pouvez créer un nouveau compte Data Lake Storage Gen2 ou en utiliser un existant. Pour en savoir plus, consultez Créer un compte de stockage à utiliser Azure Data Lake Storage.
Pour plus de cohérence, utilisez la structure de dossier suivante pour créer le raccourci :
Files\External\Imaging\DICOM\[Namespace]\[BYOSShortcutName]. La valeurNamespaceassure la séparation logique des raccourcis provenant de différents systèmes sources. Par exemple, vous pouvez utiliser le nom Data Lake Storage Gen2 pour la valeurNamespace.Note
Les raccourcis OneLake prennent également en charge plusieurs systèmes de stockage au-delà de Data Lake Storage Gen2. Pour obtenir la liste complète des types de stockage pris en charge, consultez Raccourcis OneLake.
Configurez l’ admin lakehouse pour activer BYOS :
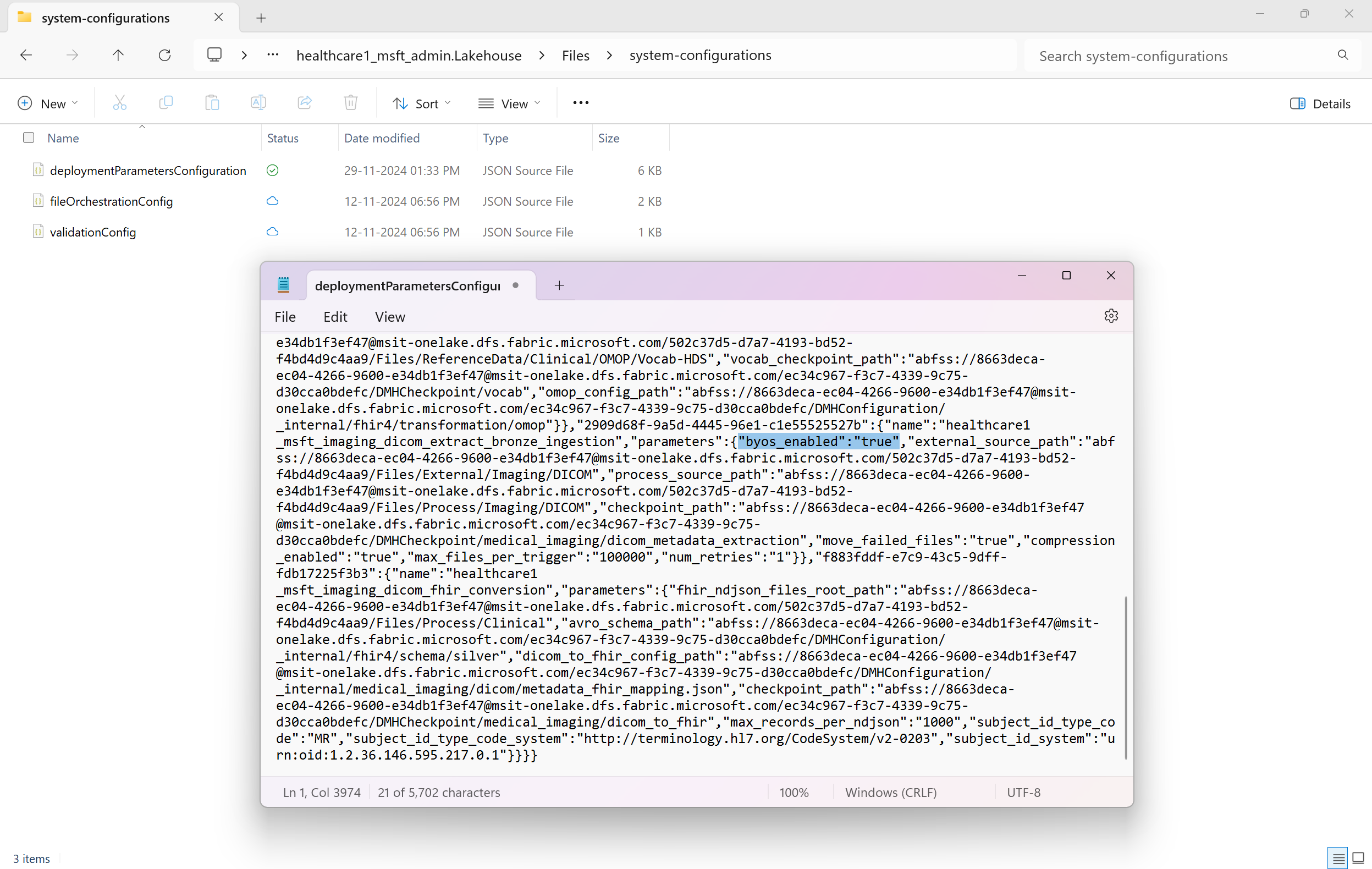
Accédez à healthcare#_msft_admin lakehouse et ouvrez le fichier deploymentParametersConfiguration.json sous
Files\system-configurations.Activez le paramètre BYOS dans ce fichier de configuration. Utilisez l’explorateur de fichiers OneLake pour ouvrir le fichier deploymentParametersConfiguration.json à partir du chemin de dossier suivant :
OneLake - Microsoft\[WorkspaceName]\healthcare#_msft_admin.Lakehouse\Files\system-configurations. Utilisez n’importe quel éditeur JSON ou de texte (tel que le Bloc-notes Windows) pour ouvrir le fichier, recherchez le paramètre et définissez-le surbyos_enabledtrue .
La capacité de transformation des données DICOM peut désormais accéder à tous vos fichiers DICOM dans leur emplacement source Data Lake Storage Gen2, quelle que soit la hiérarchie/structure du dossier. Vous n’avez pas besoin d’ingérer manuellement les fichiers DICOM en tant que Terminé dans l’option Ingérer . Commencez l’exécution à partir de étape 2 : exécutez le pipeline de données d’imagerie dans la section précédente pour utiliser le pipeline d’imagerie et transformer vos données DICOM.
Note
Pour comprendre les limites d’intégration avec le service DICOM Health Data Services Azure, consultez Intégration avec le service DICOM.