Selvstudium: Find relationer i Synthea datasæt ved hjælp af semantisk link
I dette selvstudium illustreres det, hvordan du registrerer relationer i det offentlige Synthea datasæt ved hjælp af semantisk link.
Når du arbejder med nye data eller arbejder uden en eksisterende datamodel, kan det være nyttigt at finde relationer automatisk. Denne relationsregistrering kan hjælpe dig med at:
- forstå modellen på et højt niveau,
- få mere indsigt under udforskning af dataanalyse,
- validere opdaterede data eller nye, indgående data og
- rene data.
Selvom relationer er kendt på forhånd, kan en søgning efter relationer hjælpe med en bedre forståelse af datamodellen eller identifikation af problemer med datakvaliteten.
I dette selvstudium starter du med et simpelt eksempel på en oprindelig plan, hvor du eksperimenterer med kun tre tabeller, så det er nemt at følge forbindelser mellem dem. Derefter viser du et mere komplekst eksempel med et større tabelsæt.
I dette selvstudium lærer du, hvordan du:
- Brug komponenterne i Python-biblioteket for semantiske links (SemPy), der understøtter integration med Power BI og hjælper med at automatisere dataanalyser. Disse komponenter omfatter:
- FabricDataFrame – en pandaslignende struktur, der er forbedret med yderligere semantiske oplysninger.
- Funktioner til at trække semantiske modeller fra et Fabric-arbejdsområde ind i din notesbog.
- Funktioner, der automatiserer registrering og visualisering af relationer i dine semantiske modeller.
- Foretag fejlfinding af processen til registrering af relationer for semantiske modeller med flere tabeller og indbyrdes afhængigheder.
Forudsætninger
Få et Microsoft Fabric-abonnement. Du kan også tilmelde dig en gratis Microsoft Fabric-prøveversion.
Log på Microsoft Fabric.
Brug oplevelsesskifteren nederst til venstre på startsiden til at skifte til Fabric.
- Vælg arbejdsområder i navigationsruden til venstre for at finde og vælge dit arbejdsområde. Dette arbejdsområde bliver dit aktuelle arbejdsområde.
Følg med i notesbogen
Notesbogen relationships_detection_tutorial.ipynb følger med dette selvstudium.
Hvis du vil åbne den medfølgende notesbog til dette selvstudium, skal du følge vejledningen i Forbered dit system til selvstudier om datavidenskab importere notesbogen til dit arbejdsområde.
Hvis du hellere vil kopiere og indsætte koden fra denne side, kan du oprette en ny notesbog.
Sørg for at vedhæfte et lakehouse til notesbogen, før du begynder at køre kode.
Konfigurer notesbogen
I dette afsnit skal du konfigurere et notesbogmiljø med de nødvendige moduler og data.
Installér
SemPyfra PyPI ved hjælp af funktionen%pipindbygget installation i notesbogen:%pip install semantic-linkUdfør den nødvendige import af SemPy-moduler, som du skal bruge senere:
import pandas as pd from sempy.samples import download_synthea from sempy.relationships import ( find_relationships, list_relationship_violations, plot_relationship_metadata )Importér pandas for at gennemtvinge en konfigurationsindstilling, der hjælper med outputformatering:
import pandas as pd pd.set_option('display.max_colwidth', None)Træk i eksempeldataene. I dette selvstudium skal du bruge Synthea datasæt af syntetiske medicinske journaler (lille version for nemheds skyld):
download_synthea(which='small')
Registrer relationer på et lille undersæt af Synthea tabeller
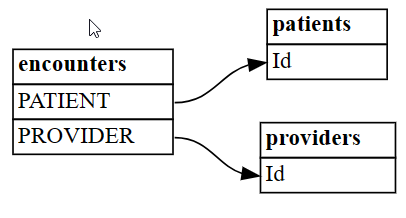
Vælg tre tabeller fra et større sæt:
-
patientsangiver patientoplysninger -
encountersangiver de patienter, der havde medicinske møder (f.eks. en lægeaftale, procedure) -
providersangiver, hvilke medicinske udbydere der deltog i patienterne
Tabellen
encountersløser en mange til mange-relation mellempatientsogprovidersog kan beskrives som et associativt objekt:patients = pd.read_csv('synthea/csv/patients.csv') providers = pd.read_csv('synthea/csv/providers.csv') encounters = pd.read_csv('synthea/csv/encounters.csv')-
Find relationer mellem tabellerne ved hjælp af SemPys
find_relationshipsfunktion:suggested_relationships = find_relationships([patients, providers, encounters]) suggested_relationshipsVisualiser relationerne DataFrame som en graf ved hjælp af SemPys
plot_relationship_metadatafunktion.plot_relationship_metadata(suggested_relationships)Funktionen viser relationshierarkiet fra venstre side til højre, hvilket svarer til tabellerne "fra" og "til" i outputtet. Med andre ord bruger de uafhængige "fra"-tabeller på venstre side deres fremmede nøgler til at pege på deres "til"-afhængighedstabeller i højre side. Hvert objektfelt viser kolonner, der deltager på enten "fra" eller "til"-siden af en relation.
Relationer genereres som standard som "m:1" (ikke som "1:m") eller "1:1". Relationerne "1:1" kan oprettes på en eller begge måder, afhængigt af om forholdet mellem tilknyttede værdier og alle værdier overskrider
coverage_thresholdi blot én eller begge retninger. Senere i dette selvstudium dækker du det mindre hyppige tilfælde af "m:m"-relationer.
Fejlfinding af problemer med registrering af relationer
Det oprindelige eksempel viser en vellykket registrering af relationer på rene Synthea- data. I praksis er dataene sjældent rene, hvilket forhindrer vellykket registrering. Der er flere teknikker, der kan være nyttige, når dataene ikke er rene.
Dette afsnit i dette selvstudium omhandler registrering af relationer, når den semantiske model indeholder ugyldige data.
Begynd med at manipulere de oprindelige DataFrames for at hente "beskadigede" data og udskrive størrelsen på de beskadigede data.
# create a dirty 'patients' dataframe by dropping some rows using head() and duplicating some rows using concat() patients_dirty = pd.concat([patients.head(1000), patients.head(50)], axis=0) # create a dirty 'providers' dataframe by dropping some rows using head() providers_dirty = providers.head(5000) # the dirty dataframes have fewer records than the clean ones print(len(patients_dirty)) print(len(providers_dirty))Til sammenligning kan du udskrive størrelser af de oprindelige tabeller:
print(len(patients)) print(len(providers))Find relationer mellem tabellerne ved hjælp af SemPys
find_relationshipsfunktion:find_relationships([patients_dirty, providers_dirty, encounters])Outputtet af koden viser, at der ikke er registreret nogen relationer på grund af de fejl, du introducerede tidligere for at oprette den "beskidte" semantiske model.
Brug validering
Validering er det bedste værktøj til fejlfinding af fejl i forbindelse med registrering af relationer, fordi:
- Den rapporterer tydeligt, hvorfor en bestemt relation ikke følger reglerne for fremmed nøgle og derfor ikke kan registreres.
- Den kører hurtigt med store semantiske modeller, fordi den kun fokuserer på de erklærede relationer og ikke udfører en søgning.
Validering kan bruge en hvilken som helst dataramme med kolonner, der ligner den, der genereres af find_relationships. I følgende kode refererer suggested_relationships DataFrame til patients i stedet for patients_dirty, men du kan aliassere DataFrames med en ordbog:
dirty_tables = {
"patients": patients_dirty,
"providers" : providers_dirty,
"encounters": encounters
}
errors = list_relationship_violations(dirty_tables, suggested_relationships)
errors
Løs søgekriterier
I mere dystre scenarier kan du prøve at løse dine søgekriterier. Denne metode øger muligheden for falske positiver.
Angiv
include_many_to_many=True, og evaluer, om det hjælper:find_relationships(dirty_tables, include_many_to_many=True, coverage_threshold=1)Resultaterne viser, at relationen fra
encounterstilpatientsblev registreret, men der er to problemer:- Relationen angiver en retning fra
patientstilencounters, som er en omvendt af den forventede relation. Dette skyldes, at allepatientser dækket afencounters(Coverage Fromer 1,0), mensencounterskun er delvist dækket afpatients(Coverage To= 0,85), da der mangler patientrækker. - Der er et utilsigtet match på en kolonne med lav kardinalitet
GENDER, som tilfældigvis matcher efter navn og værdi i begge tabeller, men det er ikke en "m:1"-relation af interesse. Den lave kardinalitet angives afUnique Count FromogUnique Count Tokolonner.
- Relationen angiver en retning fra
Kør
find_relationshipsigen for kun at søge efter "m:1"-relationer, men med et laverecoverage_threshold=0.5:find_relationships(dirty_tables, include_many_to_many=False, coverage_threshold=0.5)Resultatet viser den korrekte retning af relationerne fra
encounterstilproviders. Relationen fraencounterstilpatientsregistreres dog ikke, fordipatientsikke er entydig, så den kan ikke være på "En"-siden af "m:1"-relationen.Løs både
include_many_to_many=Trueogcoverage_threshold=0.5:find_relationships(dirty_tables, include_many_to_many=True, coverage_threshold=0.5)Nu er begge interessante relationer synlige, men der er meget mere støj:
- Den lave kardinalitetsmatch på
GENDERer til stede. - Der blev vist et match med højere kardinalitet "m:m" på
ORGANIZATIONviste, hvilket gør det tydeligt, atORGANIZATIONsandsynligvis er en kolonne, der ikke er normaliseret for begge tabeller.
- Den lave kardinalitetsmatch på
Søg efter kolonnenavne
Som standard betragter SemPy som kun attributter, der viser navnelighed, og udnytter det faktum, at databasedesignere normalt navngiver relaterede kolonner på samme måde. Denne funktionsmåde hjælper med at undgå falske relationer, der oftest forekommer med heltalsnøgler med lav kardinalitet. Hvis der f.eks. er 1,2,3,...,10 produktkategorier og 1,2,3,...,10 ordrestatuskode, forveksles de med hinanden, når du kun ser på værditilknytninger uden at tage kolonnenavne i betragtning. Falske relationer bør ikke være et problem med GUID-lignende nøgler.
SemPy ser på en lighed mellem kolonnenavne og tabelnavne. Matchning er omtrentlig, og der skelnes ikke mellem store og små bogstaver. Den ignorerer de understrenge, der oftest findes i "dekoratøren", f.eks. "id", "kode", "navn", "nøgle", "pk", "fk". Som et resultat heraf er de mest typiske matchcases:
- en attribut med navnet 'column' i enheden 'foo' svarer til en attribut med navnet 'column' (også 'COLUMN' eller 'Column') på enheden 'bar'.
- en attribut med navnet 'column' i enheden 'foo' matcher med en attribut med navnet 'column_id' på 'bar'.
- en attribut med navnet 'bar' i enheden 'foo' matcher med en attribut med navnet 'code' i 'bar'.
Ved først at matche kolonnenavne kører registreringen hurtigere.
Søg efter kolonnenavnene:
- Hvis du vil vide, hvilke kolonner der er valgt til yderligere evaluering, skal du bruge indstillingen
verbose=2(verbose=1viser kun de objekter, der behandles). - Parameteren
name_similarity_thresholdbestemmer, hvordan kolonner sammenlignes. Grænsen på 1 angiver, at du kun er interesseret i 100% match.
find_relationships(dirty_tables, verbose=2, name_similarity_threshold=1.0);Hvis du kører med 100% lighed, kan der ikke tages højde for små forskelle mellem navne. I dit eksempel har tabellerne en flertalsform med suffikset "s", hvilket ikke resulterer i et nøjagtigt match. Dette håndteres godt sammen med standard
name_similarity_threshold=0.8.- Hvis du vil vide, hvilke kolonner der er valgt til yderligere evaluering, skal du bruge indstillingen
Kør igen med standard
name_similarity_threshold=0.8:find_relationships(dirty_tables, verbose=2, name_similarity_threshold=0.8);Bemærk, at id'et for flertalsformen
patientsnu sammenlignes med entals-patientuden at føje for mange andre falske sammenligninger til udførelsestiden.Kør igen med standard
name_similarity_threshold=0:find_relationships(dirty_tables, verbose=2, name_similarity_threshold=0);Ændring af
name_similarity_thresholdtil 0 er den anden yderlighed, og det angiver, at du vil sammenligne alle kolonner. Dette er sjældent nødvendigt og resulterer i øget udførelsestid og falske match, der skal gennemgås. Se antallet af sammenligninger i det detaljerede output.
Oversigt over tip til fejlfinding
- Start fra nøjagtigt match for "m:1"-relationer (dvs. standard
include_many_to_many=Falseogcoverage_threshold=1.0). Det er normalt det, du vil have. - Brug et smalt fokus på mindre undersæt af tabeller.
- Brug validering til at registrere problemer med datakvaliteten.
- Brug
verbose=2, hvis du vil forstå, hvilke kolonner der tages i betragtning for relationen. Dette kan resultere i en stor mængde output. - Vær opmærksom på afvejninger af søgeargumenter.
include_many_to_many=Trueogcoverage_threshold<1.0kan medføre falske relationer, der kan være sværere at analysere og skal filtreres.
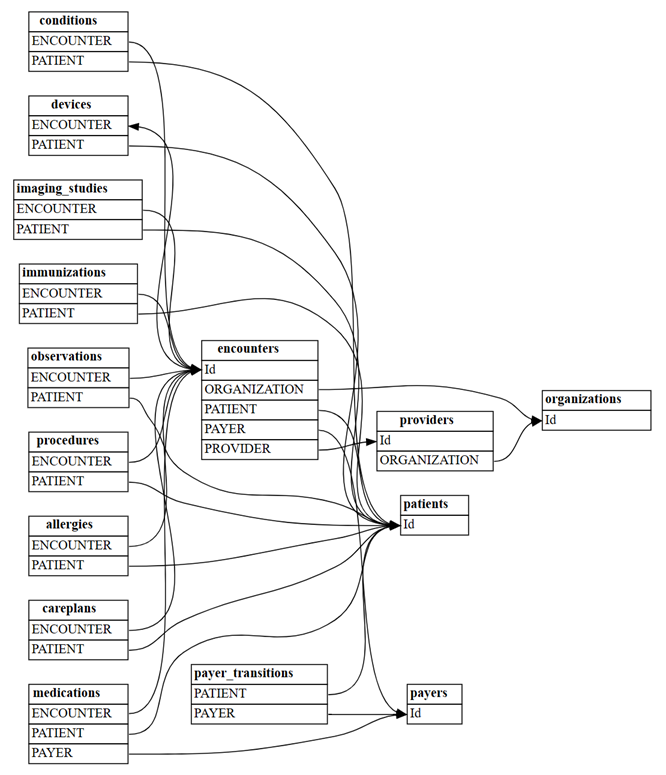
Registrer relationer på det fulde Synthea datasæt
Det enkle grundlinjeeksempel var et praktisk lærings- og fejlfindingsværktøj. I praksis kan du starte fra en semantisk model, f.eks. den fulde Synthea datasæt, som har mange flere tabeller. Udforsk den fulde synthea datasæt på følgende måde.
Læs alle filer fra mappen synthea/csv:
all_tables = { "allergies": pd.read_csv('synthea/csv/allergies.csv'), "careplans": pd.read_csv('synthea/csv/careplans.csv'), "conditions": pd.read_csv('synthea/csv/conditions.csv'), "devices": pd.read_csv('synthea/csv/devices.csv'), "encounters": pd.read_csv('synthea/csv/encounters.csv'), "imaging_studies": pd.read_csv('synthea/csv/imaging_studies.csv'), "immunizations": pd.read_csv('synthea/csv/immunizations.csv'), "medications": pd.read_csv('synthea/csv/medications.csv'), "observations": pd.read_csv('synthea/csv/observations.csv'), "organizations": pd.read_csv('synthea/csv/organizations.csv'), "patients": pd.read_csv('synthea/csv/patients.csv'), "payer_transitions": pd.read_csv('synthea/csv/payer_transitions.csv'), "payers": pd.read_csv('synthea/csv/payers.csv'), "procedures": pd.read_csv('synthea/csv/procedures.csv'), "providers": pd.read_csv('synthea/csv/providers.csv'), "supplies": pd.read_csv('synthea/csv/supplies.csv'), }Find relationer mellem tabellerne ved hjælp af SemPys
find_relationshipsfunktion:suggested_relationships = find_relationships(all_tables) suggested_relationshipsVisualiser relationer:
plot_relationship_metadata(suggested_relationships)Tæl, hvor mange nye "m:m"-relationer der registreres med
include_many_to_many=True. Disse relationer er ud over de tidligere viste "m:1"-relationer. Derfor skal du filtrere eftermultiplicity:suggested_relationships = find_relationships(all_tables, coverage_threshold=1.0, include_many_to_many=True) suggested_relationships[suggested_relationships['Multiplicity']=='m:m']Du kan sortere relationsdataene efter forskellige kolonner for at få en dybere forståelse af deres karakter. Du kan f.eks. vælge at sortere outputtet efter
Row Count FromogRow Count To, som hjælper med at identificere de største tabeller.suggested_relationships.sort_values(['Row Count From', 'Row Count To'], ascending=False)I en anden semantisk model er det måske vigtigt at fokusere på antallet af null-værdier
Null Count FromellerCoverage To.Denne analyse kan hjælpe dig med at forstå, om nogen af relationerne kan være ugyldige, og om du har brug for at fjerne dem fra listen over kandidater.
Relateret indhold
Tjek andre selvstudier for semantisk link / SemPy: