Tutorial: Descobrir relações no conjunto de dados do Synthea usando o link semântico
Este tutorial ilustra como detectar relações no conjunto de dados do Synthea público usando o link semântico.
Quando você está trabalhando com novos dados ou trabalhando sem um modelo de dados existente, pode ser útil descobrir relações automaticamente. Essa detecção de relação pode ajudá-lo a:
- entender o modelo em um alto nível
- obter mais insights durante a análise de dados exploratória,
- validar dados atualizados ou novos dados de entrada e
- e limpar dados.
Mesmo que as relações sejam conhecidas com antecedência, uma pesquisa por relações pode ajudar a entender melhor o modelo de dados ou a identificação de problemas de qualidade de dados.
Neste tutorial, você começa com um exemplo de linha de base simples em que você experimenta apenas três tabelas para que as conexões entre elas sejam fáceis de seguir. Em seguida, você mostra um exemplo mais complexo com um conjunto de tabelas maior.
Neste tutorial, você aprenderá a:
- Use componentes da biblioteca Python do link semântico (SemPy) que dão suporte à integração com o Power BI e ajudam a automatizar a análise de dados. Esses componentes incluem:
- FabricDataFrame - uma estrutura semelhante a pandas aprimorada com informações semânticas adicionais.
- Funções para extrair modelos semânticos de um workspace do Fabric para seu notebook.
- Funções que automatizam a descoberta e visualização de relações em seus modelos semânticos.
- Solucionar problemas do processo de descoberta de relação para modelos semânticos com várias tabelas e interdependências.
Pré-requisitos
Obtenha uma assinatura do Microsoft Fabric. Ou inscreva-se para uma avaliação gratuita do Microsoft Fabric.
Entre no Microsoft Fabric.
Use o seletor de experiência no canto inferior esquerdo da sua página inicial para alternar para o Fabric.
- Selecione Workspaces no painel de navegação esquerdo para localizar e selecionar seu workspace. Essa área de trabalho se torna sua área de trabalho atual.
Acompanhar no notebook
O notebook relationships_detection_tutorial.ipynb acompanha este tutorial.
Para abrir o bloco de anotações que acompanha este tutorial, siga as instruções em Preparar seu sistema para tutoriais de ciência de dados para importar o bloco de anotações para seu espaço de trabalho.
Se você prefere copiar e colar o código desta página, pode criar um notebook.
Certifique-se de anexar um lakehouse ao notebook antes de começar a executar o código.
Configurar o notebook
Nesta seção, você configurará um ambiente de notebook com os módulos e dados necessários.
Instale
SemPydo PyPI usando o recurso de instalação%pipem linha dentro do notebook:%pip install semantic-linkExecute as importações necessárias de módulos SemPy que você precisará mais tarde:
import pandas as pd from sempy.samples import download_synthea from sempy.relationships import ( find_relationships, list_relationship_violations, plot_relationship_metadata )Importe pandas para impor uma opção de configuração que ajuda na formatação de saída:
import pandas as pd pd.set_option('display.max_colwidth', None)Extraia os dados de exemplo. Para este tutorial, você usará o conjunto de dados do Synthea de registros médicos sintéticos (versão pequena para simplificar):
download_synthea(which='small')
Detectar relações em um pequeno subconjunto de tabelas Synthea
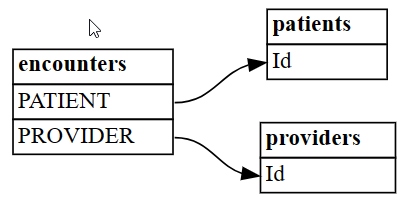
Selecione três tabelas de um conjunto maior:
patientsespecifica informações do pacienteencountersespecifica os pacientes que tiveram encontros médicos (por exemplo, uma consulta médica, procedimento)providersespecifica quais provedores médicos atenderam aos pacientes
A tabela
encountersresolve uma relação muitos para muitos entrepatientseproviderse pode ser descrita como uma entidade associativa:patients = pd.read_csv('synthea/csv/patients.csv') providers = pd.read_csv('synthea/csv/providers.csv') encounters = pd.read_csv('synthea/csv/encounters.csv')Encontre relações entre as tabelas usando a função
find_relationshipsdo SemPy:suggested_relationships = find_relationships([patients, providers, encounters]) suggested_relationshipsVisualize as relações dataframe como um grafo, usando a função
plot_relationship_metadatado SemPy.plot_relationship_metadata(suggested_relationships)A função estabelece a hierarquia de relação do lado esquerdo para o direito, o que corresponde a tabelas "de" e "para" na saída. Em outras palavras, as tabelas independentes "de" no lado esquerdo usam suas chaves de referência para apontar para suas tabelas de dependência "para" no lado direito. Cada caixa de entidade mostra colunas que participam do lado "de" ou "para" de uma relação.
Por padrão, as relações são geradas como "m:1" (não como "1:m") ou "1:1". As relações "1:1" podem ser geradas de uma ou ambas as maneiras, dependendo de a razão entre valores mapeados e todos os valores exceder
coverage_thresholdem apenas uma ou ambas as direções. Posteriormente neste tutorial, você abordará o caso menos frequente de relações "m:m".
Solucionar problemas de detecção de relação
O exemplo de linha de base mostra uma detecção de relação bem-sucedida em dados limpos Synthea. Na prática, os dados raramente são limpos, o que impede a detecção bem-sucedida. Há várias técnicas que podem ser úteis quando os dados não estão limpos.
Esta seção deste tutorial aborda a detecção de relação quando o modelo semântico contém dados sujos.
Comece manipulando os DataFrames originais para obter dados "sujos" e imprima o tamanho dos dados sujos.
# create a dirty 'patients' dataframe by dropping some rows using head() and duplicating some rows using concat() patients_dirty = pd.concat([patients.head(1000), patients.head(50)], axis=0) # create a dirty 'providers' dataframe by dropping some rows using head() providers_dirty = providers.head(5000) # the dirty dataframes have fewer records than the clean ones print(len(patients_dirty)) print(len(providers_dirty))Para comparação, imprima os tamanhos das tabelas originais:
print(len(patients)) print(len(providers))Encontre relações entre as tabelas usando a função
find_relationshipsdo SemPy:find_relationships([patients_dirty, providers_dirty, encounters])A saída do código mostra que não há relações detectadas devido aos erros que você introduziu anteriormente para criar o modelo semântico "sujo".
Usar a validação
A validação é a melhor ferramenta para solucionar falhas na detecção de relações porque:
- Ele explica claramente por que uma relação específica não segue as regras de chave estrangeira e, portanto, não pode ser detectada.
- Ele é executado rapidamente com modelos semânticos grandes porque se concentra apenas nas relações declaradas e não executa uma pesquisa.
A validação pode usar qualquer DataFrame com colunas semelhantes à gerada por find_relationships. No código a seguir, o DataFrame suggested_relationships refere-se a patients em vez de patients_dirty, mas você pode alias os DataFrames com um dicionário:
dirty_tables = {
"patients": patients_dirty,
"providers" : providers_dirty,
"encounters": encounters
}
errors = list_relationship_violations(dirty_tables, suggested_relationships)
errors
Afrouxar critérios de pesquisa
Em cenários mais obscuros, você pode tentar afrouxar seus critérios de pesquisa. Esse método aumenta a possibilidade de falsos positivos.
Defina
include_many_to_many=Truee avalie se ele ajuda:find_relationships(dirty_tables, include_many_to_many=True, coverage_threshold=1)Os resultados mostram que a relação de
encountersapatientsfoi detectada, mas há dois problemas:- A relação indica uma direção de
patientsparaencounters, que é um inverso da relação esperada. Isso ocorre porque todos ospatientspassaram a ser cobertor porencounters(Coverage Fromé 1,0), enquanto osencounterssão apenas parcialmente cobertos porpatients(Coverage To= 0,85), uma vez que as linhas dos pacientes estão ausentes. - Há uma correspondência acidental em uma coluna
GENDERde baixa cardinalidade, que acaba correspondendo por nome e valor em ambas as tabelas, mas não é uma relação de interesse "m:1". A baixa cardinalidade é indicada por colunasUnique Count FromeUnique Count To.
- A relação indica uma direção de
Execute novamente
find_relationshipspara procurar apenas relações "m:1", mas com umcoverage_threshold=0.5inferior:find_relationships(dirty_tables, include_many_to_many=False, coverage_threshold=0.5)O resultado mostra a direção correta das relações de
encountersparaproviders. No entanto, a relação deencountersapatientsnão é detectada, porquepatientsnão é exclusivo, portanto, não pode estar no lado "Um" da relação "m:1".Amenizar
include_many_to_many=Trueecoverage_threshold=0.5:find_relationships(dirty_tables, include_many_to_many=True, coverage_threshold=0.5)Agora, ambas as relações de interesse são visíveis, mas há muito mais ruído:
- A baixa correspondência de cardinalidade de
GENDERestá presente. - Apareceu uma maior correspondência de cardinalidade "m:m" de
ORGANIZATION, tornando evidente queORGANIZATIONé provavelmente uma coluna desnormalizada para ambas as tabelas.
- A baixa correspondência de cardinalidade de
Corresponder nomes de coluna
Por padrão, o SemPy considera como correspondência apenas os atributos que mostram similaridade de nome, aproveitando o fato de que os designers de banco de dados geralmente nomeam colunas relacionadas da mesma maneira. Esse comportamento ajuda a evitar relações espúrias, que ocorrem com mais frequência com chaves inteiros de baixa cardinalidade. Por exemplo, se houver 1,2,3,...,10 categorias de produto e 1,2,3,...,10 códigos de status de pedido, eles serão confundidos entre si quando apenas mapeamentos de valores forem analisados, sem levar em conta os nomes de coluna. Relações espúrias não devem ser um problema com chaves do tipo GUID.
O SemPy analisa uma semelhança entre nomes de coluna e nomes de tabela. A correspondência é aproximada e não diferencia maiúsculas de minúsculas. Ela ignora as subcadeias de caracteres "decorador" encontradas com mais frequência, como "id", "code", "name", "key", "pk" e "fk". Como resultado, os casos de correspondência mais típicos são:
- um atributo chamado “column” na entidade “foo” corresponde a um atributo chamado “column” (também “COLUMN” ou “Column”) na entidade 'bar'.
- Um atributo chamado 'column' na entidade 'foo' corresponde a um atributo chamado 'column_id' na entidade 'bar'.
- um atributo chamado “bar” na entidade “foo” corresponde a um atributo chamado “code” em “bar”.
Ao corresponder primeiro os nomes de coluna, a detecção é executada mais rapidamente.
Combine os nomes das colunas.
- Para entender quais colunas são selecionadas para avaliação adicional, use a opção
verbose=2(verbose=1lista apenas as entidades que estão sendo processadas). - O parâmetro
name_similarity_thresholddetermina como as colunas são comparadas. O limite de 1 indica que seu interesse é em apenas 100% de correspondência.
find_relationships(dirty_tables, verbose=2, name_similarity_threshold=1.0);A execução com 100% de similaridade não contabiliza pequenas diferenças entre os nomes. Em seu exemplo, as tabelas têm um formato plural com sufixo "s", o que não resulta em correspondência exata. Isso é bem resolvido com o padrão
name_similarity_threshold=0.8.- Para entender quais colunas são selecionadas para avaliação adicional, use a opção
Executar novamente com o
name_similarity_threshold=0.8padrão:find_relationships(dirty_tables, verbose=2, name_similarity_threshold=0.8);Observe que o ID para a forma plural
patientsagora é comparado aopatientsingular, sem adicionar muitas outras comparações espúrias ao tempo de execução.Executar novamente com o
name_similarity_threshold=0padrão:find_relationships(dirty_tables, verbose=2, name_similarity_threshold=0);Alterar
name_similarity_thresholdpara 0 é o outro extremo e indica que você deseja comparar todas as colunas. Isso raramente é necessário e resulta em maior tempo de execução e correspondências espúrias que precisam ser revisadas. Observe o número de comparações na saída detalhada.
Resumo das dicas de solução de problemas
- Comece a partir da correspondência exata para relações "m:1" (ou seja, o padrão
include_many_to_many=Falseecoverage_threshold=1.0). Isso geralmente é o que você quer. - Use um foco estreito em subconjuntos menores de tabelas.
- Use a validação para detectar problemas de qualidade de dados.
- Use
verbose=2se quiser entender quais colunas são consideradas para a relação. Isso pode resultar em uma grande quantidade de saída. - Esteja ciente das desvantagens dos argumentos de pesquisa.
include_many_to_many=Trueecoverage_threshold<1.0podem produzir relações espúrias que podem ser mais difíceis de analisar e precisarão ser filtradas.
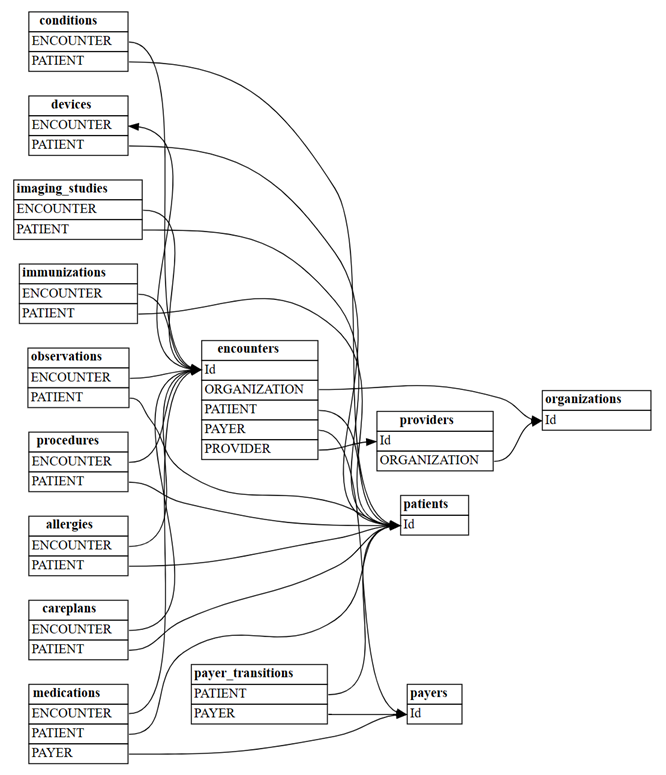
Detectar relações no conjunto de dados completo Synthea
O exemplo de linha de base simples foi uma ferramenta conveniente de aprendizado e solução de problemas. Na prática, você pode começar a partir de um modelo semântico, como o conjunto de dados de Synthea completo, que tem muito mais tabelas. Explore o conjunto de dados synthea completo da seguinte maneira.
Leia todos os arquivos do diretório synthea/csv:
all_tables = { "allergies": pd.read_csv('synthea/csv/allergies.csv'), "careplans": pd.read_csv('synthea/csv/careplans.csv'), "conditions": pd.read_csv('synthea/csv/conditions.csv'), "devices": pd.read_csv('synthea/csv/devices.csv'), "encounters": pd.read_csv('synthea/csv/encounters.csv'), "imaging_studies": pd.read_csv('synthea/csv/imaging_studies.csv'), "immunizations": pd.read_csv('synthea/csv/immunizations.csv'), "medications": pd.read_csv('synthea/csv/medications.csv'), "observations": pd.read_csv('synthea/csv/observations.csv'), "organizations": pd.read_csv('synthea/csv/organizations.csv'), "patients": pd.read_csv('synthea/csv/patients.csv'), "payer_transitions": pd.read_csv('synthea/csv/payer_transitions.csv'), "payers": pd.read_csv('synthea/csv/payers.csv'), "procedures": pd.read_csv('synthea/csv/procedures.csv'), "providers": pd.read_csv('synthea/csv/providers.csv'), "supplies": pd.read_csv('synthea/csv/supplies.csv'), }Encontre relações entre as tabelas usando a função
find_relationshipsdo SemPy:suggested_relationships = find_relationships(all_tables) suggested_relationshipsVisualizar relações:
plot_relationship_metadata(suggested_relationships)Conte quantas novas relações "m:m" serão descobertas com
include_many_to_many=True. Essas relações são além das relações "m:1" mostradas anteriormente; portanto, você precisa filtrar emmultiplicity:suggested_relationships = find_relationships(all_tables, coverage_threshold=1.0, include_many_to_many=True) suggested_relationships[suggested_relationships['Multiplicity']=='m:m']Você pode classificar os dados de relação por várias colunas para obter uma compreensão mais profunda de sua natureza. Por exemplo, você pode optar por ordenar a saída por
Row Count FromeRow Count To, que ajudam a identificar as maiores tabelas.suggested_relationships.sort_values(['Row Count From', 'Row Count To'], ascending=False)Em um modelo semântico diferente, talvez seja importante se concentrar no número de nulos
Null Count FromouCoverage To.Essa análise pode ajudá-lo a entender se alguma das relações pode ser inválida e se você precisar removê-las da lista de candidatos.
Conteúdo relacionado
Confira outros tutoriais para link semântico/SemPy: