Zelfstudie: Relaties ontdekken in de Synthea-gegevensset met behulp van een semantische koppeling
In deze zelfstudie ziet u hoe u relaties in de openbare Synthea-gegevensset detecteert met behulp van een semantische koppeling.
Wanneer u met nieuwe gegevens werkt of zonder een bestaand gegevensmodel werkt, kan het handig zijn om automatisch relaties te detecteren. Deze relatiedetectie kan u helpen bij het volgende:
- het model op hoog niveau begrijpen,
- krijg meer inzichten tijdens verkennende gegevensanalyse,
- bijgewerkte gegevens of nieuwe, binnenkomende gegevens valideren en
- gegevens opschonen.
Zelfs als relaties van tevoren bekend zijn, kan een zoekopdracht naar relaties helpen bij een beter begrip van het gegevensmodel of het identificeren van problemen met de kwaliteit van gegevens.
In deze zelfstudie begint u met een eenvoudig basislijnvoorbeeld waarin u met slechts drie tabellen experimenteert, zodat verbindingen tussen deze tabellen eenvoudig te volgen zijn. Vervolgens geeft u een complexer voorbeeld weer met een grotere tabelset.
In deze zelfstudie leert u het volgende:
- Gebruik onderdelen van de Python-bibliotheek (SemPy) van semantische koppelingen die ondersteuning bieden voor integratie met Power BI en om gegevensanalyse te automatiseren. Deze onderdelen zijn onder andere:
- FabricDataFrame- een pandas-achtige structuur die is uitgebreid met aanvullende semantische informatie.
- Functies voor het ophalen van semantische modellen uit een Fabric-werkruimte in uw notebook.
- Functies waarmee de detectie en visualisatie van relaties in uw semantische modellen wordt geautomatiseerd.
- Los het proces van relatiedetectie voor semantische modellen met meerdere tabellen en afhankelijkheden op.
Vereisten
Haal een Microsoft Fabric-abonnement op. Of meld u aan voor een gratis proefversie van Microsoft Fabric.
Meld u aan bij Microsoft Fabric.
Gebruik de ervaringswisselaar aan de linkerkant van de startpagina om over te schakelen naar de Synapse-Datawetenschap-ervaring.

- Selecteer Werkruimten in het linkernavigatiedeelvenster om uw werkruimte te zoeken en te selecteren. Deze werkruimte wordt uw huidige werkruimte.
Volgen in het notitieblok
De notebook relationships_detection_tutorial.ipynb begeleidt deze zelfstudie.
Als u het bijbehorende notitieblok voor deze zelfstudie wilt openen, volgt u de instructies in Uw systeem voorbereiden voor zelfstudies voor gegevenswetenschap om het notebook te importeren in uw werkruimte.
Als u liever de code van deze pagina kopieert en plakt, kunt u een nieuw notitieblok maken.
Zorg ervoor dat u een lakehouse aan het notebook koppelt voordat u begint met het uitvoeren van code.
Het notebook instellen
In deze sectie stelt u een notebookomgeving in met de benodigde modules en gegevens.
Installeren
SemPyvanuit PyPI met behulp van de%pipinline-installatiemogelijkheid in het notebook:%pip install semantic-linkVoer de benodigde importbewerkingen uit van SemPy-modules die u later nodig hebt:
import pandas as pd from sempy.samples import download_synthea from sempy.relationships import ( find_relationships, list_relationship_violations, plot_relationship_metadata )Importeer pandas voor het afdwingen van een configuratieoptie die helpt bij het opmaken van uitvoer:
import pandas as pd pd.set_option('display.max_colwidth', None)Haal de voorbeeldgegevens op. Voor deze zelfstudie gebruikt u de Synthea-gegevensset van synthetische medische records (kleine versie voor eenvoud):
download_synthea(which='small')
Relaties detecteren op een kleine subset van Synthea-tabellen
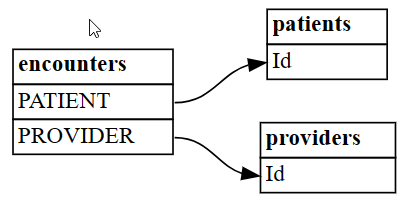
Selecteer drie tabellen uit een grotere set:
patientsgeeft patiëntinformatie opencountersgeeft de patiënten aan die medische ontmoeting hadden (bijvoorbeeld een medische afspraak, procedure)providersgeeft aan welke medische dienstverleners aan de patiënten zijn deelgenomen
De
encounterstabel lost een veel-op-veel-relatie tussenpatientsenproviderskan worden beschreven als een associatieve entiteit:patients = pd.read_csv('synthea/csv/patients.csv') providers = pd.read_csv('synthea/csv/providers.csv') encounters = pd.read_csv('synthea/csv/encounters.csv')Zoek relaties tussen de tabellen met behulp van de functie van SemPy
find_relationships:suggested_relationships = find_relationships([patients, providers, encounters]) suggested_relationshipsVisualiseer de relaties dataframe als een grafiek met behulp van de functie van
plot_relationship_metadataSemPy.plot_relationship_metadata(suggested_relationships)Met de functie wordt de relatiehiërarchie aan de linkerkant aan de rechterkant beschreven, die overeenkomt met tabellen van 'van' en 'naar' in de uitvoer. Met andere woorden, de onafhankelijke 'from'-tabellen aan de linkerkant gebruiken hun refererende sleutels om te verwijzen naar hun 'naar'-afhankelijkheidstabellen aan de rechterkant. Elk entiteitsvak bevat kolommen die deelnemen aan de 'van' of 'naar'-kant van een relatie.
Standaard worden relaties gegenereerd als 'm:1' (niet als '1:m') of '1:1'. De relaties 1:1 kunnen op een of beide manieren worden gegenereerd, afhankelijk van of de verhouding tussen toegewezen waarden en alle waarden in slechts één of beide richtingen
coverage_thresholdwordt overschreden. Verderop in deze zelfstudie behandelt u het minder frequente geval van 'm:m'-relaties.
Problemen met relatiedetectie oplossen
In het basislijnvoorbeeld ziet u een geslaagde relatiedetectie voor schone Synthea-gegevens . In de praktijk worden de gegevens zelden opgeschoond, waardoor succesvolle detectie wordt voorkomen. Er zijn verschillende technieken die nuttig kunnen zijn wanneer de gegevens niet worden opgeschoond.
In deze sectie van deze zelfstudie wordt de detectie van relaties opgelost wanneer het semantische model vuile gegevens bevat.
Begin met het bewerken van de oorspronkelijke DataFrames om 'vuile' gegevens te verkrijgen en de grootte van de vuile gegevens af te drukken.
# create a dirty 'patients' dataframe by dropping some rows using head() and duplicating some rows using concat() patients_dirty = pd.concat([patients.head(1000), patients.head(50)], axis=0) # create a dirty 'providers' dataframe by dropping some rows using head() providers_dirty = providers.head(5000) # the dirty dataframes have fewer records than the clean ones print(len(patients_dirty)) print(len(providers_dirty))Ter vergelijking: afdrukgrootten van de oorspronkelijke tabellen:
print(len(patients)) print(len(providers))Zoek relaties tussen de tabellen met behulp van de functie van SemPy
find_relationships:find_relationships([patients_dirty, providers_dirty, encounters])In de uitvoer van de code ziet u dat er geen relaties zijn gedetecteerd vanwege de fouten die u eerder hebt geïntroduceerd om het semantische model 'vuil' te maken.
Validatie gebruiken
Validatie is het beste hulpprogramma voor het oplossen van fouten in relatiedetectie, omdat:
- Het rapporteert duidelijk waarom een bepaalde relatie niet aan de regels voor refererende sleutels wordt gevolgd en daarom niet kan worden gedetecteerd.
- Het wordt snel uitgevoerd met grote semantische modellen, omdat deze zich alleen richt op de gedeclareerde relaties en geen zoekopdracht uitvoert.
Validatie kan elk DataFrame gebruiken met kolommen die vergelijkbaar zijn met de kolommen die zijn gegenereerd door find_relationships. In de volgende code verwijst het suggested_relationships DataFrame naar patients in plaats patients_dirtyvan , maar u kunt de DataFrames aliasen met een woordenlijst:
dirty_tables = {
"patients": patients_dirty,
"providers" : providers_dirty,
"encounters": encounters
}
errors = list_relationship_violations(dirty_tables, suggested_relationships)
errors
Losse zoekcriteria
In meer duistere scenario's kunt u proberen uw zoekcriteria los te maken. Deze methode verhoogt de kans op fout-positieven.
Instellen
include_many_to_many=Trueen evalueren of dit helpt:find_relationships(dirty_tables, include_many_to_many=True, coverage_threshold=1)De resultaten laten zien dat de relatie van
encountersnaar ispatientsgedetecteerd, maar er zijn twee problemen:- De relatie geeft een richting aan van
patientsnaarencounters, wat een omgekeerde van de verwachte relatie is. Dit komt doordat allespatientsis gedektencountersdoor (Coverage Fromis 1,0) terwijlencountersslechts gedeeltelijk wordt gedekt doorpatients(Coverage To= 0,85), omdat patiëntenrijen ontbreken. - Er is een onbedoelde overeenkomst op een kolom met lage kardinaliteit
GENDER, die overeenkomt met naam en waarde in beide tabellen, maar het is geen 'm:1'-relatie van belang. De lage kardinaliteit wordt aangegeven doorUnique Count FromenUnique Count Tokolommen.
- De relatie geeft een richting aan van
Voer
find_relationshipsopnieuw uit om alleen te zoeken naar 'm:1'-relaties, maar met een lagerecoverage_threshold=0.5:find_relationships(dirty_tables, include_many_to_many=False, coverage_threshold=0.5)Het resultaat toont de juiste richting van de relaties van
encounterstotproviders. De relatie vanencountersnaarpatientswordt echter niet gedetecteerd, omdatpatientsdeze niet uniek is, dus deze kan niet aan de 'een'-kant van de relatie 'm:1' staan.Los zowel
include_many_to_many=Truecoverage_threshold=0.5als :find_relationships(dirty_tables, include_many_to_many=True, coverage_threshold=0.5)Nu zijn beide relaties van belang zichtbaar, maar er is veel meer ruis:
- De lage kardinaliteitsmatch
GENDERis aanwezig. - Er wordt een hogere kardinaliteit 'm:m'
ORGANIZATIONweergegeven, waardoor het duidelijk is datORGANIZATIONeen kolom die niet is genormaliseerd voor beide tabellen.
- De lage kardinaliteitsmatch
Kolomnamen vergelijken
SemPy beschouwt standaard als overeenkomsten met alleen kenmerken die overeenkomen met naamovereenkomsten, waarbij ze profiteren van het feit dat databaseontwerpers meestal gerelateerde kolommen op dezelfde manier noemen. Dit gedrag helpt bij het voorkomen van valse relaties, die het vaakst voorkomen met gehele getallen met een lage kardinaliteit. Als er bijvoorbeeld productcategorieën en 1,2,3,...,10 orderstatuscode zijn1,2,3,...,10, worden ze met elkaar verward wanneer alleen waardetoewijzingen worden bekeken zonder rekening te houden met kolomnamen. Valse relaties mogen geen probleem zijn met GUID-achtige sleutels.
SemPy kijkt naar een overeenkomst tussen kolomnamen en tabelnamen. De overeenkomst is bij benadering en niet hoofdlettergevoelig. De subtekenreeksen van de decorator worden genegeerd, zoals 'id', 'code', 'name', 'key', 'pk', 'fk'. Als gevolg hiervan zijn de meest voorkomende overeenkomstcases:
- een kenmerk met de naam 'kolom' in entiteit 'foo' komt overeen met een kenmerk met de naam 'column' (ook 'COLUMN' of 'Column') in de entiteit 'bar'.
- een kenmerk met de naam 'kolom' in entiteit 'foo' komt overeen met een kenmerk met de naam 'column_id' in 'bar'.
- een kenmerk met de naam 'bar' in entiteit 'foo' komt overeen met een kenmerk met de naam 'code' in 'bar'.
Door eerst de kolomnamen te vergelijken, wordt de detectie sneller uitgevoerd.
Overeenkomen met de kolomnamen:
- Als u wilt weten welke kolommen zijn geselecteerd voor verdere evaluatie, gebruikt u de
verbose=2optie (verbose=1alleen de entiteiten die worden verwerkt). - De
name_similarity_thresholdparameter bepaalt hoe kolommen worden vergeleken. De drempelwaarde van 1 geeft aan dat u alleen geïnteresseerd bent in een overeenkomst van 100%.
find_relationships(dirty_tables, verbose=2, name_similarity_threshold=1.0);Bij een overeenkomst van 100% kan geen rekening worden gehouden met kleine verschillen tussen namen. In uw voorbeeld hebben de tabellen een meervoudsvorm met achtervoegsel 's', wat resulteert in geen exacte overeenkomst. Dit is goed afgehandeld met de standaard
name_similarity_threshold=0.8.- Als u wilt weten welke kolommen zijn geselecteerd voor verdere evaluatie, gebruikt u de
Opnieuw uitvoeren met de standaardwaarde
name_similarity_threshold=0.8:find_relationships(dirty_tables, verbose=2, name_similarity_threshold=0.8);U ziet dat de id voor het meervoudformulier
patientsnu wordt vergeleken met de enkelvoudpatientzonder te veel andere valse vergelijkingen toe te voegen aan de uitvoeringstijd.Opnieuw uitvoeren met de standaardwaarde
name_similarity_threshold=0:find_relationships(dirty_tables, verbose=2, name_similarity_threshold=0);Overstappen op
name_similarity_threshold0 is het andere extreme en geeft aan dat u alle kolommen wilt vergelijken. Dit is zelden nodig en resulteert in een verhoogde uitvoeringstijd en valse overeenkomsten die moeten worden gecontroleerd. Bekijk het aantal vergelijkingen in de uitgebreide uitvoer.
Overzicht van tips voor probleemoplossing
- Begin met exacte overeenkomst voor 'm:1'-relaties (dat wil zeggen, de standaard
include_many_to_many=Falseencoverage_threshold=1.0). Dit is meestal wat u wilt. - Gebruik een beperkte focus op kleinere subsets van tabellen.
- Gebruik validatie om problemen met gegevenskwaliteit te detecteren.
- Gebruik
verbose=2deze optie als u wilt weten welke kolommen in aanmerking komen voor relatie. Dit kan leiden tot een grote hoeveelheid uitvoer. - Let op de afwegingen van zoekargumenten.
include_many_to_many=Trueencoverage_threshold<1.0kan ongewenste relaties produceren die moeilijker te analyseren zijn en moeten worden gefilterd.
Relaties detecteren op de volledige Synthea-gegevensset
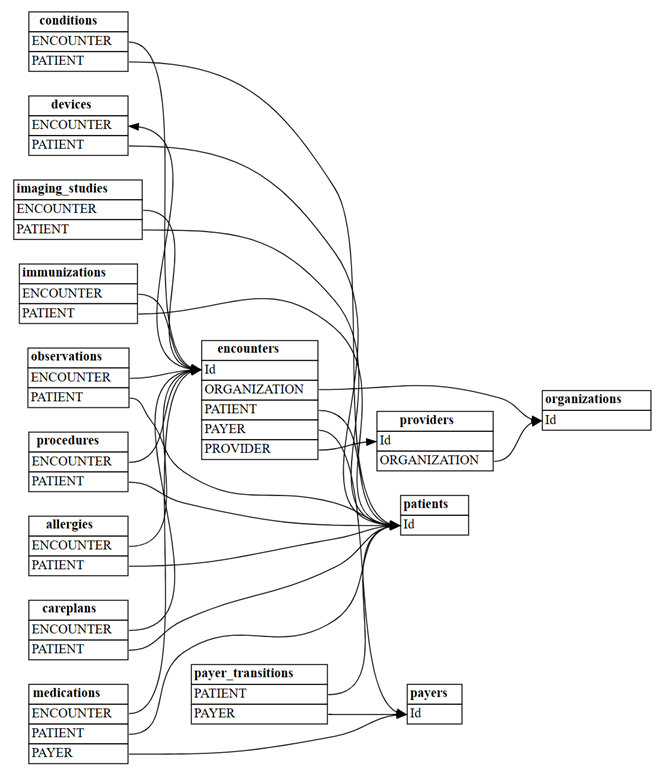
Het eenvoudige basislijnvoorbeeld was een handig leer- en probleemoplossingsprogramma. In de praktijk kunt u beginnen met een semantisch model, zoals de volledige Synthea-gegevensset , die veel meer tabellen bevat. Verken de volledige synthea-gegevensset als volgt.
Alle bestanden uit de map synthea/csv lezen:
all_tables = { "allergies": pd.read_csv('synthea/csv/allergies.csv'), "careplans": pd.read_csv('synthea/csv/careplans.csv'), "conditions": pd.read_csv('synthea/csv/conditions.csv'), "devices": pd.read_csv('synthea/csv/devices.csv'), "encounters": pd.read_csv('synthea/csv/encounters.csv'), "imaging_studies": pd.read_csv('synthea/csv/imaging_studies.csv'), "immunizations": pd.read_csv('synthea/csv/immunizations.csv'), "medications": pd.read_csv('synthea/csv/medications.csv'), "observations": pd.read_csv('synthea/csv/observations.csv'), "organizations": pd.read_csv('synthea/csv/organizations.csv'), "patients": pd.read_csv('synthea/csv/patients.csv'), "payer_transitions": pd.read_csv('synthea/csv/payer_transitions.csv'), "payers": pd.read_csv('synthea/csv/payers.csv'), "procedures": pd.read_csv('synthea/csv/procedures.csv'), "providers": pd.read_csv('synthea/csv/providers.csv'), "supplies": pd.read_csv('synthea/csv/supplies.csv'), }Zoek relaties tussen de tabellen met behulp van de functie van
find_relationshipsSemPy:suggested_relationships = find_relationships(all_tables) suggested_relationshipsRelaties visualiseren:
plot_relationship_metadata(suggested_relationships)Tel met hoeveel nieuwe 'm:m'-relaties worden gedetecteerd
include_many_to_many=True. Deze relaties zijn naast de eerder getoonde 'm:1'-relaties; Daarom moet u filteren opmultiplicity:suggested_relationships = find_relationships(all_tables, coverage_threshold=1.0, include_many_to_many=True) suggested_relationships[suggested_relationships['Multiplicity']=='m:m']U kunt de relatiegegevens sorteren op verschillende kolommen om meer inzicht te krijgen in hun aard. U kunt er bijvoorbeeld voor kiezen om de uitvoer
Row Count Fromte orden enRow Count To, waarmee u de grootste tabellen kunt identificeren.suggested_relationships.sort_values(['Row Count From', 'Row Count To'], ascending=False)In een ander semantisch model zou het misschien belangrijk zijn om te focussen op het aantal null-waarden
Null Count FromofCoverage To.Deze analyse kan u helpen te begrijpen of een van de relaties ongeldig kan zijn en of u ze uit de lijst met kandidaten moet verwijderen.
Gerelateerde inhoud
Bekijk andere zelfstudies voor semantische koppeling /SemPy:
- Zelfstudie: Gegevens opschonen met functionele afhankelijkheden
- Zelfstudie: Functionele afhankelijkheden analyseren in een semantisch voorbeeldmodel
- Zelfstudie: Relaties ontdekken in een semantisch model met behulp van een semantische koppeling
- Zelfstudie: Power BI-metingen extraheren en berekenen uit een Jupyter-notebook