Opplæring: Oppdag relasjoner i Synthea-datasettet ved hjelp av semantisk kobling
Denne opplæringen illustrerer hvordan du oppdager relasjoner i det offentlige Synthea-datasettet ved hjelp av semantisk kobling.
Når du arbeider med nye data eller arbeider uten en eksisterende datamodell, kan det være nyttig å oppdage relasjoner automatisk. Denne relasjonsgjenkjenningen kan hjelpe deg med å:
- forstå modellen på et høyt nivå,
- få mer innsikt under utforskende dataanalyse,
- validere oppdaterte data eller nye, innkommende data og
- rengjøre data.
Selv om relasjoner er kjent på forhånd, kan et søk etter relasjoner hjelpe med bedre forståelse av datamodellen eller identifisering av problemer med datakvalitet.
I denne opplæringen begynner du med et enkelt eksempel på grunnlinje der du eksperimenterer med bare tre tabeller, slik at tilkoblinger mellom dem er enkle å følge. Deretter viser du et mer komplekst eksempel med et større tabellsett.
I denne opplæringen lærer du hvordan du kan gjøre følgende:
- Bruk komponenter i Python-biblioteket for semantiske koblinger (SemPy) som støtter integrering med Power BI, og hjelp til å automatisere dataanalyse. Disse komponentene omfatter:
- FabricDataFrame - en pandas-lignende struktur forbedret med ekstra semantisk informasjon.
- Funksjoner for å trekke semantiske modeller fra et Fabric-arbeidsområde inn i notatblokken.
- Funksjoner som automatiserer oppdagelse og visualisering av relasjoner i semantiske modeller.
- Feilsøk prosessen med relasjonsgjenkjenning for semantiske modeller med flere tabeller og interdependencies.
Forutsetning
Få et Microsoft Fabric-abonnement. Eller registrer deg for en gratis prøveversjon av Microsoft Fabric.
Logg på Microsoft Fabric.
Bruk opplevelsesbryteren til venstre på hjemmesiden for å bytte til Synapse Data Science-opplevelsen.

- Velg Arbeidsområder fra venstre navigasjonsrute for å finne og velge arbeidsområdet. Dette arbeidsområdet blir ditt gjeldende arbeidsområde.
Følg med i notatblokken
Notatblokken relationships_detection_tutorial.ipynb følger med denne opplæringen.
Hvis du vil åpne den medfølgende notatblokken for denne opplæringen, følger du instruksjonene i Klargjør systemet for opplæring for datavitenskap, for å importere notatblokken til arbeidsområdet.
Hvis du heller vil kopiere og lime inn koden fra denne siden, kan du opprette en ny notatblokk.
Pass på at du fester et lakehouse til notatblokken før du begynner å kjøre kode.
Konfigurere notatblokken
I denne delen konfigurerer du et notatblokkmiljø med de nødvendige modulene og dataene.
Installer
SemPyfra PyPI ved hjelp av%pipden innebygde installasjonsfunksjonen i notatblokken:%pip install semantic-linkUtfør nødvendige importer av SemPy-moduler som du trenger senere:
import pandas as pd from sempy.samples import download_synthea from sempy.relationships import ( find_relationships, list_relationship_violations, plot_relationship_metadata )Importer pandaer for å fremtvinge et konfigurasjonsalternativ som hjelper med utdataformatering:
import pandas as pd pd.set_option('display.max_colwidth', None)Hente eksempeldataene. I denne opplæringen bruker du Synthea-datasettet for syntetiske medisinske journaler (liten versjon for enkelhetsbruk):
download_synthea(which='small')
Oppdage relasjoner i et lite delsett av Synthea-tabeller
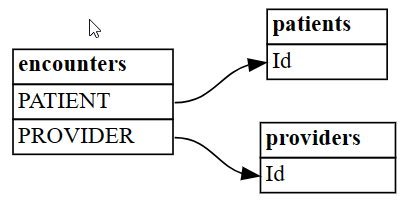
Velg tre tabeller fra et større sett:
patientsangir pasientinformasjonencountersangir pasientene som hadde medisinske møter (for eksempel en medisinsk avtale, prosedyre)providersangir hvilke medisinske leverandører som deltok på pasientene
Tabellen
encountersløser en mange-til-mange-relasjon mellompatientsogproviderskan beskrives som en tilknyttet enhet:patients = pd.read_csv('synthea/csv/patients.csv') providers = pd.read_csv('synthea/csv/providers.csv') encounters = pd.read_csv('synthea/csv/encounters.csv')Finn relasjoner mellom tabellene ved hjelp av SemPys
find_relationshipsfunksjon:suggested_relationships = find_relationships([patients, providers, encounters]) suggested_relationshipsVisualiser relasjonene DataFrame som en graf ved hjelp av SemPys
plot_relationship_metadatafunksjon.plot_relationship_metadata(suggested_relationships)Funksjonen angir relasjonshierarkiet fra venstre side til høyre, som tilsvarer «fra» og «til»-tabeller i utdataene. De uavhengige «fra»-tabellene på venstre side bruker med andre ord sekundærnøklene til å peke til avhengighetstabellene «til» på høyre side. Hver enhetsboks viser kolonner som deltar enten på «fra»- eller «til»-siden i en relasjon.
Som standard genereres relasjoner som «m:1» (ikke som «1:m») eller «1:1». Relasjonene 1:1 kan genereres på én eller begge måter, avhengig av om forholdet mellom tilordnede verdier og alle verdier overskrider
coverage_thresholdi bare én eller begge retninger. Senere i denne opplæringen dekker du det sjeldnere tilfellet av «m:m»-relasjoner.
Feilsøke problemer med gjenkjenning av relasjoner
Det opprinnelige eksemplet viser en vellykket relasjonsgjenkjenning på rene Synthea-data . I praksis er dataene sjelden rene, noe som hindrer vellykket gjenkjenning. Det finnes flere teknikker som kan være nyttige når dataene ikke er rene.
Denne delen av denne opplæringen tar for seg gjenkjenning av relasjoner når den semantiske modellen inneholder skitne data.
Begynn med å manipulere de opprinnelige DataFrames for å hente "skitne" data, og skriv ut størrelsen på de skitne dataene.
# create a dirty 'patients' dataframe by dropping some rows using head() and duplicating some rows using concat() patients_dirty = pd.concat([patients.head(1000), patients.head(50)], axis=0) # create a dirty 'providers' dataframe by dropping some rows using head() providers_dirty = providers.head(5000) # the dirty dataframes have fewer records than the clean ones print(len(patients_dirty)) print(len(providers_dirty))Til sammenligning skriver du ut størrelsen på de opprinnelige tabellene:
print(len(patients)) print(len(providers))Finn relasjoner mellom tabellene ved hjelp av SemPys
find_relationshipsfunksjon:find_relationships([patients_dirty, providers_dirty, encounters])Utdataene for koden viser at det ikke er oppdaget noen relasjoner på grunn av feilene du introduserte tidligere for å opprette den "skitne" semantiske modellen.
Bruk validering
Validering er det beste verktøyet for feilsøking av gjenkjenningsfeil for relasjon fordi:
- Den rapporterer tydelig hvorfor en bestemt relasjon ikke følger sekundærnøkkelreglene og derfor ikke kan oppdages.
- Den kjører raskt med store semantiske modeller fordi den bare fokuserer på de deklarerte relasjonene og ikke utfører et søk.
Validering kan bruke en dataramme med kolonner som ligner på den som genereres av find_relationships. I følgende kode suggested_relationships refererer DataFrame til patients i stedet patients_dirtyfor , men du kan alias datarammene med en ordliste:
dirty_tables = {
"patients": patients_dirty,
"providers" : providers_dirty,
"encounters": encounters
}
errors = list_relationship_violations(dirty_tables, suggested_relationships)
errors
Løsne søkekriterier
I mer skumle scenarioer kan du prøve å løsne søkekriteriene. Denne metoden øker muligheten for falske positiver.
Angi
include_many_to_many=Trueog evaluer om det hjelper:find_relationships(dirty_tables, include_many_to_many=True, coverage_threshold=1)Resultatene viser at relasjonen fra
encounterstilpatientsble oppdaget, men det er to problemer:- Relasjonen angir en retning fra
patientstilencounters, som er en omvendt av den forventede relasjonen. Dette er fordi altpatientsskjedde dekket avencounters(Coverage Fromer 1,0) mensencounterser bare delvis dekket avpatients(Coverage To= 0,85), siden pasienter rader mangler. - Det er et tilfeldig treff på en kolonne med lav kardinalitet
GENDER, som tilfeldigvis samsvarer med navn og verdi i begge tabellene, men det er ikke en «m:1»-relasjon av interesse. Den lave kardinaliteten angis avUnique Count FromogUnique Count Tokolonner.
- Relasjonen angir en retning fra
Kjør på
find_relationshipsnytt for å se bare etter "m:1"-relasjoner, men med en laverecoverage_threshold=0.5:find_relationships(dirty_tables, include_many_to_many=False, coverage_threshold=0.5)Resultatet viser riktig retning av relasjonene fra
encounterstilproviders. Relasjonen fraencounterstilpatientsoppdages imidlertid ikke, fordipatientsden ikke er unik, så den kan ikke være på «Én»-siden av «m:1»-relasjonen.Løsne begge
include_many_to_many=Truedeler ogcoverage_threshold=0.5:find_relationships(dirty_tables, include_many_to_many=True, coverage_threshold=0.5)Nå er begge relasjoner av interesse synlige, men det er mye mer støy:
- Den lave kardinaliteten samsvarer på
GENDERer til stede. - En høyere kardinalitet "m:m" match på
ORGANIZATIONdukket opp, noe som gjør det tydelig atORGANIZATIONer sannsynlig en kolonne de-normalisert til begge tabellene.
- Den lave kardinaliteten samsvarer på
Samsvare kolonnenavn
Som standard anser SemPy som samsvarer bare med attributter som viser navneanfall, og drar nytte av det faktum at databaseutformere vanligvis navngir relaterte kolonner på samme måte. Denne virkemåten bidrar til å unngå falske relasjoner, som forekommer oftest med heltallsnøkler med lav kardinalitet. Hvis det for eksempel finnes 1,2,3,...,10 produktkategorier og 1,2,3,...,10 ordrestatuskode, blir de forvekslet med hverandre når de bare ser på verditilordninger uten å ta hensyn til kolonnenavn. Falske relasjoner bør ikke være et problem med GUID-lignende nøkler.
SemPy ser på en likhet mellom kolonnenavn og tabellnavn. Samsvaret er omtrentlig og skilles ikke mellom store og små bokstaver. Den ignorerer de vanligste understrengene «dekoratør», for eksempel «id», «kode», «navn», «nøkkel», «pk», «fk». Som et resultat er de mest vanlige samsvarstilfellene:
- et attributt kalt "kolonne" i enhetens foo samsvarer med et attributt kalt "kolonne" (også "KOLONNE" eller "Kolonne") i enhetens stolpe.
- et attributt kalt «kolonne» i enhetens «foo» samsvarer med et attributt kalt «column_id» i «stolpe».
- et attributt kalt «stolpe» i enhetens «foo» samsvarer med et attributt kalt «kode» i «strek».
Ved å samsvare kolonnenavn først kjører gjenkjenningen raskere.
Samsvar med kolonnenavnene:
- Hvis du vil forstå hvilke kolonner som er valgt for videre evaluering, bruker
verbose=2du alternativet (verbose=1viser bare enhetene som behandles). - Parameteren
name_similarity_thresholdbestemmer hvordan kolonner sammenlignes. Terskelen på 1 indikerer at du bare er interessert i 100 % samsvar.
find_relationships(dirty_tables, verbose=2, name_similarity_threshold=1.0);Å kjøre med 100 % likhet kan ikke ta hensyn til små forskjeller mellom navn. I eksemplet ditt har tabellene et flertallsform med suffiks, noe som resulterer i ingen nøyaktig samsvar. Dette håndteres godt med standard
name_similarity_threshold=0.8.- Hvis du vil forstå hvilke kolonner som er valgt for videre evaluering, bruker
Kjør på nytt med standard:
name_similarity_threshold=0.8find_relationships(dirty_tables, verbose=2, name_similarity_threshold=0.8);Legg merke til at ID-en for flertallsform
patientsnå sammenlignes med entallpatientuten å legge til for mange andre falske sammenligninger av utførelsestiden.Kjør på nytt med standard:
name_similarity_threshold=0find_relationships(dirty_tables, verbose=2, name_similarity_threshold=0);Endring
name_similarity_thresholdtil 0 er den andre ytterligheten, og den angir at du vil sammenligne alle kolonner. Dette er sjeldent nødvendig og resulterer i økt utførelsestid og falske kamper som må gjennomgås. Se antall sammenligninger i de detaljerte utdataene.
Sammendrag av feilsøkingstips
- Start fra nøyaktig samsvar for «m:1»-relasjoner (det vil eksempel: standard
include_many_to_many=Falseogcoverage_threshold=1.0). Dette er vanligvis det du vil ha. - Bruk et smalt fokus på mindre delsett av tabeller.
- Bruk validering til å oppdage problemer med datakvalitet.
- Bruk
verbose=2hvis du vil forstå hvilke kolonner som vurderes for relasjon. Dette kan resultere i en stor mengde utdata. - Vær oppmerksom på avveininger av søkeargumenter.
include_many_to_many=Trueogcoverage_threshold<1.0kan produsere falske relasjoner som kan være vanskeligere å analysere og må filtreres.
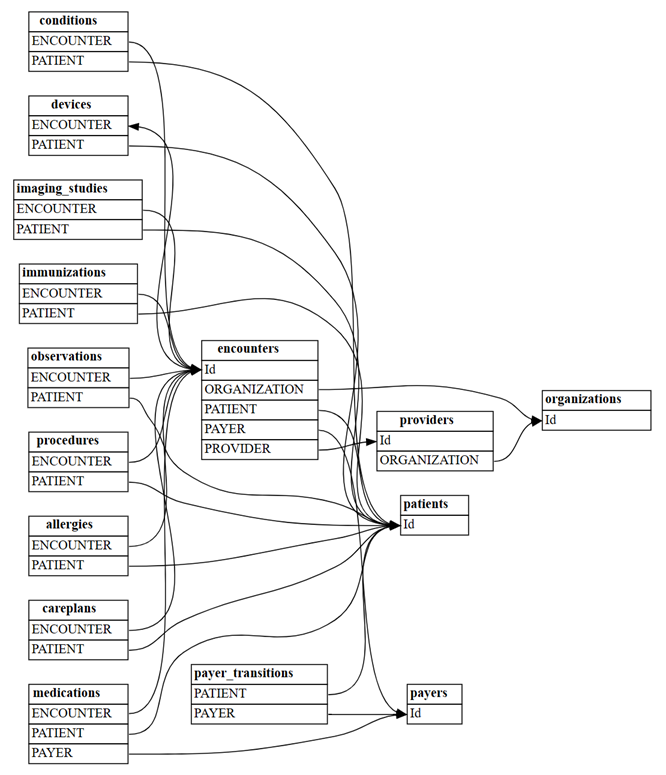
Oppdage relasjoner på det fullstendige Synthea-datasettet
Det enkle eksemplet på grunnlinje var et praktisk verktøy for læring og feilsøking. I praksis kan du starte fra en semantisk modell, for eksempel det fullstendige Synthea-datasettet , som har mange flere tabeller. Utforsk det fullstendige synthea-datasettet på følgende måte.
Les alle filer fra synthea/csv-katalogen :
all_tables = { "allergies": pd.read_csv('synthea/csv/allergies.csv'), "careplans": pd.read_csv('synthea/csv/careplans.csv'), "conditions": pd.read_csv('synthea/csv/conditions.csv'), "devices": pd.read_csv('synthea/csv/devices.csv'), "encounters": pd.read_csv('synthea/csv/encounters.csv'), "imaging_studies": pd.read_csv('synthea/csv/imaging_studies.csv'), "immunizations": pd.read_csv('synthea/csv/immunizations.csv'), "medications": pd.read_csv('synthea/csv/medications.csv'), "observations": pd.read_csv('synthea/csv/observations.csv'), "organizations": pd.read_csv('synthea/csv/organizations.csv'), "patients": pd.read_csv('synthea/csv/patients.csv'), "payer_transitions": pd.read_csv('synthea/csv/payer_transitions.csv'), "payers": pd.read_csv('synthea/csv/payers.csv'), "procedures": pd.read_csv('synthea/csv/procedures.csv'), "providers": pd.read_csv('synthea/csv/providers.csv'), "supplies": pd.read_csv('synthea/csv/supplies.csv'), }Finn relasjoner mellom tabellene ved hjelp av SemPys
find_relationshipsfunksjon:suggested_relationships = find_relationships(all_tables) suggested_relationshipsVisualiser relasjoner:
plot_relationship_metadata(suggested_relationships)Tell hvor mange nye «m:m»-relasjoner som vil bli oppdaget med
include_many_to_many=True. Disse relasjonene er i tillegg til de tidligere viste «m:1»-relasjonene. Derfor må du filtrere påmultiplicity:suggested_relationships = find_relationships(all_tables, coverage_threshold=1.0, include_many_to_many=True) suggested_relationships[suggested_relationships['Multiplicity']=='m:m']Du kan sortere relasjonsdataene etter ulike kolonner for å få en dypere forståelse av deres natur. Du kan for eksempel velge å bestille utdataene etter
Row Count FromogRow Count To, som bidrar til å identifisere de største tabellene.suggested_relationships.sort_values(['Row Count From', 'Row Count To'], ascending=False)I en annen semantisk modell er det kanskje viktig å fokusere på antall nullverdier
Null Count FromellerCoverage To.Denne analysen kan hjelpe deg med å forstå om noen av relasjonene kan være ugyldige, og om du trenger å fjerne dem fra listen over kandidater.
Relatert innhold
Sjekk ut andre opplæringer for semantisk kobling / SemPy: