Självstudie: Identifiera relationer i datauppsättningen Synthea med hjälp av semantisk länk
Den här handledningen visar hur du identifierar relationer i den offentliga Synthea-datauppsättningen med hjälp av semantiska länkar.
När du arbetar med nya data eller arbetar utan en befintlig datamodell kan det vara bra att identifiera relationer automatiskt. Den här relationsidentifieringen kan hjälpa dig att:
- förstå modellen på en hög nivå,
- få fler insikter under undersökande dataanalys,
- verifiera uppdaterade data eller nya, inkommande data och
- rensa data.
Även om relationer är kända i förväg kan en sökning efter relationer hjälpa till med bättre förståelse av datamodellen eller identifiering av datakvalitetsproblem.
I den här självstudien börjar du med ett enkelt baslinjeexempel där du bara experimenterar med tre tabeller så att anslutningar mellan dem är lätta att följa. Sedan visar du ett mer komplext exempel med en större tabelluppsättning.
I den här handledningen lär du dig:
- Använd komponenter i semantiklänkens Python-bibliotek (SemPy) som stöder integrering med Power BI och hjälper till att automatisera dataanalys. Dessa komponenter omfattar:
- FabricDataFrame – en Pandas-liknande struktur som utökas med ytterligare semantisk information.
- Funktioner för att hämta semantiska modeller från en Fabric-arbetsyta till din notebook.
- Funktioner som automatiserar identifiering och visualisering av relationer i dina semantiska modeller.
- Felsöka processen för relationsidentifiering för semantiska modeller med flera tabeller och beroenden.
Förutsättningar
Skaffa en Microsoft Fabric-prenumeration. Eller registrera dig för en kostnadsfri Microsoft Fabric-utvärderingsversion.
Logga in på Microsoft Fabric.
Använd upplevelseväxlaren längst ned till vänster på startsidan för att växla till Fabric.

- Välj Arbetsytor i det vänstra navigeringsfönstret för att hitta och välja din arbetsyta. Den här arbetsytan blir din aktuella arbetsyta.
Följ med i anteckningsboken
Den relationships_detection_tutorial.ipynb notebook-filen medföljer den här handledningen.
För att öppna den medföljande anteckningsboken för den här självstudien följer du anvisningarna i Förbered ditt system för datavetenskapliga självstudier för att importera anteckningsboken till din arbetsyta.
Om du hellre kopierar och klistrar in koden från den här sidan kan du skapa en ny notebook-fil.
Se till att ansluta en lakehouse till anteckningsboken innan du börjar köra kod.
Konfigurera anteckningsboken
I det här avsnittet konfigurerar du en notebook-miljö med nödvändiga moduler och data.
Installera
SemPyfrån PyPI med hjälp av%pip:s möjlighet till in-line-installation i notebooken.%pip install semantic-linkUtför nödvändiga importer av SemPy-moduler som du behöver senare:
import pandas as pd from sempy.samples import download_synthea from sempy.relationships import ( find_relationships, list_relationship_violations, plot_relationship_metadata )Importera Pandas för att framtvinga ett konfigurationsalternativ som hjälper till med utdataformatering:
import pandas as pd pd.set_option('display.max_colwidth', None)Hämta exempeldata. I den här självstudien använder du Synthea datauppsättning för syntetiska medicinska journaler (liten version för enkelhetens skull):
download_synthea(which='small')
Identifiera relationer i en liten delmängd av Synthea tabeller
Välj tre tabeller från en större uppsättning:
-
patientsanger patientinformationen -
encountersanger de patienter som hade medicinska möten (till exempel en medicinsk tid, procedur) -
providersanger vilka medicinska vårdgivare som tog hand om patienterna.
Tabellen
encounterslöser en många-till-många-relation mellanpatientsochprovidersoch kan beskrivas som en associativ entitet:patients = pd.read_csv('synthea/csv/patients.csv') providers = pd.read_csv('synthea/csv/providers.csv') encounters = pd.read_csv('synthea/csv/encounters.csv')-
Hitta relationer mellan tabellerna med hjälp av SemPys
find_relationshipsfunktion:suggested_relationships = find_relationships([patients, providers, encounters]) suggested_relationshipsVisualisera relationerna DataFrame som ett diagram med hjälp av SemPys
plot_relationship_metadatafunktion.plot_relationship_metadata(suggested_relationships)Funktionen beskriver relationshierarkin från vänster sida till höger, vilket motsvarar tabellerna "från" och "till" i utdata. Med andra ord använder de oberoende "från"-tabellerna på vänster sida sina sekundärnycklar för att peka på beroendetabellerna "till" till höger. Varje entitetsruta visar kolumner som deltar antingen på "från"-sidan eller "till"-sidan i en relation.
Som standard genereras relationer som "m:1" (inte som "1:m") eller "1:1". Relationerna "1:1" kan genereras på ett eller båda sätten, beroende på om förhållandet mellan mappade värden och alla värden överskrider
coverage_thresholdi bara en eller båda riktningarna. Senare i den här handledningen tar du upp det mindre vanliga fallet med "m:m"-relationer.
Felsöka problem med relationsidentifiering
Baslinjeexemplet visar en lyckad relationsidentifiering på ren Synthea data. I praktiken är data sällan rena, vilket förhindrar lyckad identifiering. Det finns flera tekniker som kan vara användbara när data inte är rena.
Det här avsnittet i den här självstudien handlar om relationsidentifiering när den semantiska modellen innehåller smutsiga data.
Börja med att ändra de ursprungliga DataFrames för att hämta "smutsiga" data och skriva ut storleken på de smutsiga data.
# create a dirty 'patients' dataframe by dropping some rows using head() and duplicating some rows using concat() patients_dirty = pd.concat([patients.head(1000), patients.head(50)], axis=0) # create a dirty 'providers' dataframe by dropping some rows using head() providers_dirty = providers.head(5000) # the dirty dataframes have fewer records than the clean ones print(len(patients_dirty)) print(len(providers_dirty))Som jämförelse kan du använda utskriftsstorlekar för de ursprungliga tabellerna:
print(len(patients)) print(len(providers))Hitta relationer mellan tabellerna med hjälp av SemPys
find_relationshipsfunktion:find_relationships([patients_dirty, providers_dirty, encounters])Kodens utdata visar att inga relationer har identifierats på grund av de fel som du introducerade tidigare för att skapa den "smutsiga" semantiska modellen.
Använd validering
Validering är det bästa verktyget för att felsöka problem med att identifiera relationer eftersom:
- Den rapporterar tydligt varför en viss relation inte följer reglerna för extern nyckel och därför inte kan upptäckas.
- Den körs snabbt med stora semantiska modeller eftersom den endast fokuserar på de deklarerade relationerna och inte utför någon sökning.
Validering kan använda valfri DataFrame med kolumner som liknar den som genereras av find_relationships. I följande kod refererar suggested_relationships DataFrame till patients i stället för patients_dirty, men du kan aliasera DataFrames med en ordlista:
dirty_tables = {
"patients": patients_dirty,
"providers" : providers_dirty,
"encounters": encounters
}
errors = list_relationship_violations(dirty_tables, suggested_relationships)
errors
Lösa upp sökvillkor
I mer skumma scenarier kan du försöka lösa upp dina sökkriterier. Den här metoden ökar risken för falska positiva identifieringar.
Ange
include_many_to_many=Trueoch utvärdera om det hjälper:find_relationships(dirty_tables, include_many_to_many=True, coverage_threshold=1)Resultaten visar att relationen från
encounterstillpatientsidentifierades, men det finns två problem:- Relationen anger en riktning från
patientstillencounters, vilket är en invertering av den förväntade relationen. Detta beror på att allapatientsråkade omfattas avencounters(Coverage Fromär 1,0) medanencountersendast delvis täcks avpatients(Coverage To= 0,85), eftersom patienternas rader saknas. - Det finns en oavsiktlig matchning på en kolumn med låg kardinalitet
GENDER, som råkar matcha efter namn och värde i båda tabellerna, men det är inte en "m:1"-relation som är av intresse. Den låga kardinaliteten anges av kolumnernaUnique Count FromochUnique Count To.
- Relationen anger en riktning från
Kör om
find_relationshipsför att bara söka efter "m:1"-relationer, men med en lägrecoverage_threshold=0.5:find_relationships(dirty_tables, include_many_to_many=False, coverage_threshold=0.5)Resultatet visar rätt riktning för relationerna från
encounterstillproviders. Relationen frånencounterstillpatientsidentifieras dock inte eftersompatientsinte är unik, så den kan inte vara på "En"-sidan av "m:1"-relationen.Lossa både
include_many_to_many=Trueochcoverage_threshold=0.5:find_relationships(dirty_tables, include_many_to_many=True, coverage_threshold=0.5)Nu är båda relationerna av intresse synliga, men det finns mycket mer brus:
- Den låga kardinalitetsmatchningen på
GENDERfinns. - En högre kardinalitetsmatchning "m:m" på
ORGANIZATIONvisades, vilket gör det uppenbart attORGANIZATIONsannolikt är en kolumn som avnormaliseras för båda tabellerna.
- Den låga kardinalitetsmatchningen på
Matcha kolumnnamn
SemPy betraktar som standard endast attribut som visar namnlikhet, vilket drar nytta av det faktum att databasdesigners vanligtvis namnger relaterade kolumner på samma sätt. Det här beteendet hjälper till att undvika falska relationer, som oftast förekommer med heltalsnycklar med låg kardinalitet. Om det till exempel finns 1,2,3,...,10 produktkategorier och 1,2,3,...,10 orderstatuskod, kommer de att förväxlas med varandra när de bara tittar på värdemappningar utan att ta hänsyn till kolumnnamn. Falska relationer ska inte vara ett problem med nycklar som liknar GUID.
SemPy tittar på en likhet mellan kolumnnamn och tabellnamn. Matchningen är ungefärlig och skiftlägesokänslig. Den ignorerar de mest förekommande "dekoratörsundersträngarna", till exempel "id", "code", "name", "key", "pk", "fk". Därför är de vanligaste matchningsfallen:
- ett attribut med namnet "column" i entiteten "foo" matchar med ett attribut som heter "column" (även "COLUMN" eller "Column") i entiteten "bar".
- ett attribut med namnet "column" i entiteten "foo" matchar med ett attribut som kallas "column_id" i "bar".
- ett attribut med namnet "bar" i entiteten "foo" matchar med ett attribut som kallas "kod" i "bar".
Genom att först matcha kolumnnamnen körs identifieringen snabbare.
Matcha kolumnnamnen:
- Om du vill förstå vilka kolumner som väljs för ytterligare utvärdering använder du alternativet
verbose=2(verbose=1endast visar de entiteter som bearbetas). - Parametern
name_similarity_thresholdavgör hur kolumner jämförs. Tröskelvärdet 1 anger att du bara är intresserad av 100% matchning.
find_relationships(dirty_tables, verbose=2, name_similarity_threshold=1.0);Om du kör med 100% kan likheten inte ta hänsyn till små skillnader mellan namn. I ditt exempel har tabellerna ett pluralform med suffixet "s", vilket inte resulterar i någon exakt matchning. Detta hanteras väl med standardvärdet
name_similarity_threshold=0.8.- Om du vill förstå vilka kolumner som väljs för ytterligare utvärdering använder du alternativet
Kör igen med standardvärdet
name_similarity_threshold=0.8:find_relationships(dirty_tables, verbose=2, name_similarity_threshold=0.8);Observera att ID för pluralform
patientsnu jämförs med singularpatientutan att lägga till för många andra oviktiga jämförelser av körningstiden.Kör igen med standardvärdet
name_similarity_threshold=0:find_relationships(dirty_tables, verbose=2, name_similarity_threshold=0);Att ändra
name_similarity_thresholdtill 0 är den andra extrem, och det anger att du vill jämföra alla kolumner. Detta är sällan nödvändigt och resulterar i ökad exekveringstid och falska matchningar som behöver granskas. Observera antalet jämförelser i utförliga utdata.
Sammanfattning av felsökningstips
- Utgå från en exakt matchning för "m:1"-relationer (det vill säga standard
include_many_to_many=Falseochcoverage_threshold=1.0). Detta är vanligtvis vad du vill. - Använd ett smalt fokus på mindre delmängder av tabeller.
- Använd validering för att identifiera datakvalitetsproblem.
- Använd
verbose=2om du vill förstå vilka kolumner som ska beaktas för relationen. Detta kan resultera i en stor mängd utdata. - Var medveten om avvägningar med sökargument.
include_many_to_many=Trueochcoverage_threshold<1.0kan skapa falska relationer som kan vara svårare att analysera och måste filtreras.
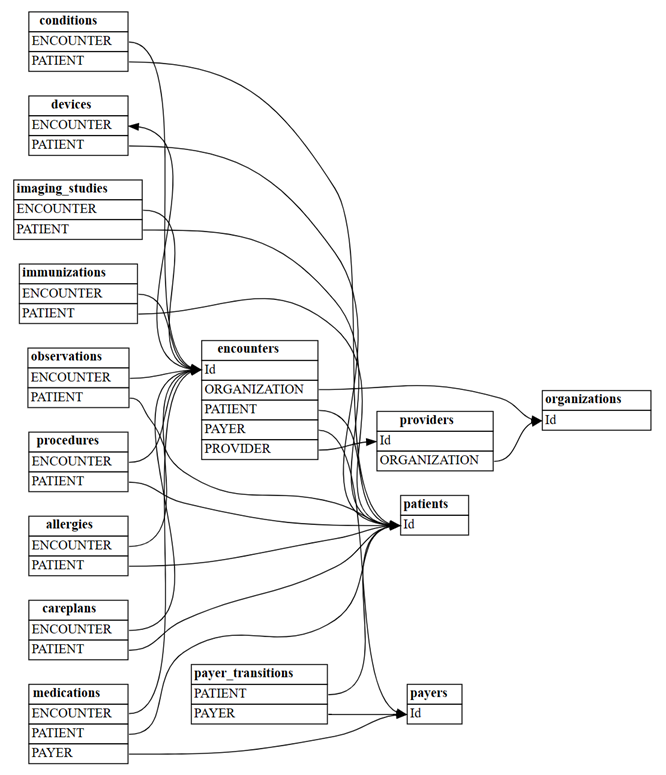
Identifiera relationer på den fullständiga Synthea-datauppsättningen
Det enkla baslinjeexemplet var ett praktiskt verktyg för inlärning och felsökning. I praktiken kan du börja från en semantisk modell, till exempel den fullständiga Synthea datauppsättning, som har många fler tabeller. Utforska hela synthea datauppsättning enligt följande.
Läs alla filer från katalogen synthea/csv:
all_tables = { "allergies": pd.read_csv('synthea/csv/allergies.csv'), "careplans": pd.read_csv('synthea/csv/careplans.csv'), "conditions": pd.read_csv('synthea/csv/conditions.csv'), "devices": pd.read_csv('synthea/csv/devices.csv'), "encounters": pd.read_csv('synthea/csv/encounters.csv'), "imaging_studies": pd.read_csv('synthea/csv/imaging_studies.csv'), "immunizations": pd.read_csv('synthea/csv/immunizations.csv'), "medications": pd.read_csv('synthea/csv/medications.csv'), "observations": pd.read_csv('synthea/csv/observations.csv'), "organizations": pd.read_csv('synthea/csv/organizations.csv'), "patients": pd.read_csv('synthea/csv/patients.csv'), "payer_transitions": pd.read_csv('synthea/csv/payer_transitions.csv'), "payers": pd.read_csv('synthea/csv/payers.csv'), "procedures": pd.read_csv('synthea/csv/procedures.csv'), "providers": pd.read_csv('synthea/csv/providers.csv'), "supplies": pd.read_csv('synthea/csv/supplies.csv'), }Hitta relationer mellan tabellerna med hjälp av SemPys
find_relationshipsfunktion:suggested_relationships = find_relationships(all_tables) suggested_relationshipsVisualisera relationer:
plot_relationship_metadata(suggested_relationships)Räkna hur många nya "m:m"-relationer som ska upptäckas med
include_many_to_many=True. Dessa relationer är utöver de tidigare visade "m:1"-relationerna. Därför måste du filtrera påmultiplicity:suggested_relationships = find_relationships(all_tables, coverage_threshold=1.0, include_many_to_many=True) suggested_relationships[suggested_relationships['Multiplicity']=='m:m']Du kan sortera relationsdata efter olika kolumner för att få en djupare förståelse för deras natur. Du kan till exempel välja att beställa utdata efter
Row Count FromochRow Count To, som hjälper dig att identifiera de största tabellerna.suggested_relationships.sort_values(['Row Count From', 'Row Count To'], ascending=False)I en annan semantisk modell kanske det är viktigt att fokusera på antalet null-värden
Null Count FromellerCoverage To.Den här analysen kan hjälpa dig att förstå om någon av relationerna kan vara ogiltiga och om du behöver ta bort dem från listan över kandidater.
Relaterat innehåll
Kolla in andra handledningar för semantiska länkar och SemPy.