Tutoriel : Découvrir les relations dans le jeu de données Synthea, à l’aide du lien sémantique
Ce tutoriel montre comment détecter les relations dans le jeu de données public Synthea en utilisant le lien sémantique.
Lorsque vous travaillez avec de nouvelles données ou que vous travaillez sans modèle de données existant, il peut être utile de découvrir automatiquement les relations. Cette détection de relation peut vous aider à :
- comprendre le modèle à un niveau élevé,
- obtenir plus d’insights lors de l’analyse exploratoire des données,
- valider les nouvelles données ou celles mises à jour, les données entrantes, et
- Données propres.
Même si les relations sont connues à l’avance, une recherche de relations peut vous aider à mieux comprendre le modèle de données ou l’identification des problèmes de qualité des données.
Dans ce tutoriel, vous commencez par un exemple de base simple où vous expérimentez seulement trois tables afin que les connexions entre elles soient faciles à suivre. Ensuite, un exemple plus complexe est affiché avec un ensemble de tables plus volumineux.
Dans ce tutoriel, vous allez apprendre à :
- Utiliser des composants de la bibliothèque Python (SemPy) du lien sémantique qui prennent en charge l’intégration à Power BI et vous aident à automatiser l’analyse des données. Ces composants sont les suivants :
- FabricDataFrame : structure de type pandas améliorée avec des informations sémantiques supplémentaires.
- Des fonctions permettant de tirer des modèles sémantiques d’un espace de travail Fabric dans votre notebook.
- Des fonctions qui automatisent la découverte et la visualisation des relations dans vos modèles sémantiques.
- Résoudre les problèmes de processus de découverte de relation pour les modèles sémantiques avec plusieurs tables et interdépendances.
Prérequis
Obtenir un abonnement Microsoft Fabric. Ou, inscrivez-vous pour un essai gratuit de Microsoft Fabric.
Connectez-vous à Microsoft Fabric.
Utilisez le sélecteur d’expérience sur le côté gauche de votre page d’accueil pour passer à l’expérience science des données Synapse.
- Sélectionnez Espaces de travail dans le volet de navigation gauche pour rechercher et sélectionner votre espace de travail. Cet espace de travail devient votre espace de travail actuel.
Suivre le notebook
Le notebook relationships_detection_tutorial.ipynb accompagne ce tutoriel.
Pour ouvrir le notebook accompagnant ce tutoriel, suivez les instructions fournies dans Préparer votre système pour le tutoriel sur la science des données afin d’importer les notebooks dans votre espace de travail.
Si vous préférez copier et coller le code de cette page, vous pouvez créer un nouveau notebook.
Assurez-vous d’attacher un lakehouse au notebook avant de commencer à exécuter du code.
Configurer le notebook
Dans cette section, vous configurez un environnement de notebook avec les modules et données nécessaires.
Installez
SemPyà partir de PyPI en utilisant la fonctionnalité d’installation inline%pipdans le notebook :%pip install semantic-linkEffectuez les importations nécessaires des modules SemPy que vous devez utiliser par la suite :
import pandas as pd from sempy.samples import download_synthea from sempy.relationships import ( find_relationships, list_relationship_violations, plot_relationship_metadata )Importez pandas pour appliquer une option de configuration qui permet de mettre en forme la sortie :
import pandas as pd pd.set_option('display.max_colwidth', None)Extrayez les exemples de données. Pour ce tutoriel, vous allez utiliser le jeu de données Synthea de dossiers médicaux synthétiques (version réduite pour plus de simplicité) :
download_synthea(which='small')
Détecter les relations sur un petit sous-ensemble de tables Synthea
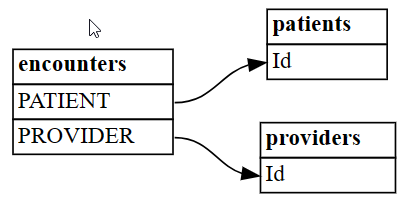
Sélectionnez trois tables dans un ensemble plus grand :
patientsspécifie les informations des patientsencountersspécifie les patients qui ont eu des rencontres médicales (par exemple, un rendez-vous médical, une procédure)providersspécifie quels prestataires médicaux ont pris en charge les patients
La table
encountersrésout une relation plusieurs-à-plusieurs entrepatientsetproviderset peut être décrite comme une entité associative :patients = pd.read_csv('synthea/csv/patients.csv') providers = pd.read_csv('synthea/csv/providers.csv') encounters = pd.read_csv('synthea/csv/encounters.csv')Recherchez des relations entre les tables à l’aide de la fonction
find_relationshipsde SemPy :suggested_relationships = find_relationships([patients, providers, encounters]) suggested_relationshipsVisualisez les relations DataFrame dans un graphique en utilisant la fonction
plot_relationship_metadatade SemPy.plot_relationship_metadata(suggested_relationships)La fonction définit la hiérarchie de relation du côté gauche vers le côté droit, qui correspond aux tables « from » et « to » dans la sortie. En d’autres termes, les tables indépendantes « from » situées à gauche utilisent leurs clés étrangères pour pointer vers leurs tables de dépendance « to » sur le côté droit. Chaque zone d’entité affiche les colonnes qui participent du côté « from » ou « to » d’une relation.
Par défaut, les relations sont générées sous la forme « m:1 » (pas comme « 1:m ») ou « 1:1 ». Les relations « 1:1 » peuvent être générées d’une ou des deux façons, selon que le ratio des valeurs mappées à toutes les valeurs dépasse
coverage_thresholddans une ou les deux directions. Plus loin dans ce tutoriel, vous découvrirez le cas moins fréquent des relations « m:m ».
Résoudre les problèmes de détection des relations
L’exemple de base montre une détection de relation réussie sur des données Synthea propres. En pratique, les données sont rarement nettoyées, ce qui empêche une détection réussie. Il existe plusieurs techniques qui peuvent être utiles lorsque les données ne sont pas nettoyées.
Cette section de ce tutoriel traite de la détection des relations lorsque le modèle sémantique contient des données non nettoyées.
Commencez par manipuler les DataFrames d’origine pour obtenir des données « sales » et imprimer la taille des données non nettoyées.
# create a dirty 'patients' dataframe by dropping some rows using head() and duplicating some rows using concat() patients_dirty = pd.concat([patients.head(1000), patients.head(50)], axis=0) # create a dirty 'providers' dataframe by dropping some rows using head() providers_dirty = providers.head(5000) # the dirty dataframes have fewer records than the clean ones print(len(patients_dirty)) print(len(providers_dirty))Pour la comparaison, imprimez les tailles des tables d’origine :
print(len(patients)) print(len(providers))Recherchez des relations entre les tables à l’aide de la fonction
find_relationshipsde SemPy :find_relationships([patients_dirty, providers_dirty, encounters])La sortie du code indique qu’aucune relation n’est détectée en raison des erreurs que vous avez introduites précédemment pour créer le modèle sémantique « non nettoyé ».
Utiliser la validation
La validation est le meilleur outil pour résoudre les échecs de détection des relations, car :
- Elle signale clairement pourquoi une relation particulière ne suit pas les règles de Clé étrangère et ne peut donc pas être détectée.
- Elle s’exécute rapidement avec des modèles sémantiques volumineux, car elle se concentre uniquement sur les relations déclarées et n’effectue pas de recherche.
La validation peut utiliser n’importe quel DataFrame avec des colonnes similaires à celles générées par find_relationships. Dans le code suivant, le DataFrame suggested_relationships fait référence à patients plutôt qu’à patients_dirty, mais vous pouvez créer un alias pour les DataFrames avec un dictionnaire :
dirty_tables = {
"patients": patients_dirty,
"providers" : providers_dirty,
"encounters": encounters
}
errors = list_relationship_violations(dirty_tables, suggested_relationships)
errors
Assouplir les critères de recherche
Dans des scénarios plus troubles, vous pouvez essayer d’assouplir vos critères de recherche. Cette méthode augmente la possibilité de faux positifs.
Définissez
include_many_to_many=Trueet évaluez si c’est utile :find_relationships(dirty_tables, include_many_to_many=True, coverage_threshold=1)Les résultats montrent que la relation entre
encountersetpatientsa été détectée, mais il existe deux problèmes :- La relation indique une direction de
patientsàencounters, ce qui est l’inverse de la relation attendue. Cela est dû au fait que tous lespatientsont été couverts par lesencounters(Coverage Fromest 1,0) alors que lesencountersne sont couverts que partiellement par lespatients(Coverage To= 0,85), car les lignes des patients sont manquantes. - Il existe une correspondance accidentelle sur une colonne
GENDERde faible cardinalité, qui correspond par nom et valeur dans les deux tables, mais il ne s’agit pas d’une relation « m:1 » d’intérêt. La cardinalité faible est indiquée par les colonnesUnique Count FrometUnique Count To.
- La relation indique une direction de
Réexécutez
find_relationshipspour rechercher uniquement les relations « m:1 », mais avec une valeurcoverage_threshold=0.5inférieure :find_relationships(dirty_tables, include_many_to_many=False, coverage_threshold=0.5)Le résultat affiche la direction correcte des relations de
encountersàproviders. Toutefois, la relation deencountersàpatientsn’est pas détectée, carpatientsn’est pas unique, elle ne peut donc pas être du côté « Un » de la relation « m:1 ».Assouplissez
include_many_to_many=Trueetcoverage_threshold=0.5:find_relationships(dirty_tables, include_many_to_many=True, coverage_threshold=0.5)Maintenant, les deux relations d’intérêt sont visibles, mais il y a beaucoup plus de d’informations :
- La correspondance de cardinalité faible sur
GENDERest présente. - Une correspondance « m:m » de cardinalité plus élevée sur
ORGANIZATIONest apparue, ce qui rend évident queORGANIZATIONest probablement une colonne dé-normalisée dans les deux tables.
- La correspondance de cardinalité faible sur
Mettre en correspondance les noms de colonnes
Par défaut, SemPy considère comme correspondance uniquement les attributs qui affichent une similarité du nom, en tirant parti du fait que les concepteurs de base de données nomment généralement les colonnes associées de la même façon. Ce comportement permet d’éviter des relations erronées, qui se produisent plus fréquemment avec des clés entières de cardinalité faible. Par exemple, s’il existe des catégories de produits 1,2,3,...,10 et un code d’état de commande 1,2,3,...,10, elles sont confondus les unes avec les autres lorsque vous examinez uniquement les mappages de valeurs sans prendre en compte les noms de colonnes. Les relations erronées ne devraient pas être un problème avec les clés de type GUID.
SemPy étudie une similarité entre les noms de colonnes et les noms de table. La correspondance est approximative et ne respecte pas la casse. Elle ignore les substrings « decorator » les plus fréquemment rencontrées, telles que « id », « code », « name », « key », « pk », « fk ». Par conséquent, les cas de correspondance les plus classiques sont les suivants :
- un attribut appelé « column » dans l’entité « foo » correspond à un attribut appelé « column » (également « COLUMN » ou « Column ») dans l’entité « bar ».
- un attribut appelé « column » dans l’entité « foo » correspond à un attribut appelé « column_id » dans « bar ».
- un attribut appelé « bar » dans l’entité « foo » correspond à un attribut appelé « code » dans « bar ».
En mettant en correspondance les noms de colonnes en premier, la détection s’exécute plus rapidement.
Mettez en correspondance les noms de colonnes :
- Pour comprendre quelles colonnes sont sélectionnées pour une évaluation ultérieure, utilisez l’option
verbose=2(verbose=1répertorie uniquement les entités en cours de traitement). - Le paramètre
name_similarity_thresholddétermine la façon dont les colonnes sont comparées. Le seuil de 1 indique que vous êtes intéressé uniquement par les correspondances à 100 %.
find_relationships(dirty_tables, verbose=2, name_similarity_threshold=1.0);L’exécution avec une similarité à 100 % ne tient pas compte des petites différences entre les noms. Dans votre exemple, les tables ont une forme au pluriel avec le suffixe « s », ce qui n’entraîne aucune correspondance exacte. Cela est géré correctement avec la valeur
name_similarity_threshold=0.8par défaut.- Pour comprendre quelles colonnes sont sélectionnées pour une évaluation ultérieure, utilisez l’option
Réexécutez avec la valeur
name_similarity_threshold=0.8par défaut :find_relationships(dirty_tables, verbose=2, name_similarity_threshold=0.8);Notez que l’ID de
patientsà la forme plurielle est maintenant comparé àpatientau singulier sans ajouter trop d’autres comparaisons erronées au temps d’exécution.Réexécutez avec la valeur
name_similarity_threshold=0par défaut :find_relationships(dirty_tables, verbose=2, name_similarity_threshold=0);La modification de
name_similarity_thresholdà 0 est l’autre extrême et indique que vous souhaitez comparer toutes les colonnes. Cela est rarement nécessaire et entraîne une augmentation du temps d’exécution et des correspondances erronées qui doivent être examinées. Observez le nombre de comparaisons dans la sortie détaillée.
Résumé des astuces de dépannage
- Commencez à partir d’une correspondance exacte pour les relations « m:1 » (autrement dit,
include_many_to_many=Falseetcoverage_threshold=1.0par défaut). C’est généralement l’option souhaitée. - Utilisez un focus étroit sur des sous-ensembles de tables plus petits.
- Utilisez la validation pour détecter les problèmes de qualité des données.
- Utilisez
verbose=2si vous souhaitez comprendre les colonnes prises en compte pour la relation. Cela peut entraîner une grande quantité de sortie. - Tenez compte des compromis des arguments de recherche.
include_many_to_many=Trueetcoverage_threshold<1.0peuvent produire des relations erronées qui peuvent être plus difficiles à analyser et qui devront être filtrées.
Détecter des relations sur le jeu de données complet Synthea
L’exemple de base simple était un outil pratique d’apprentissage et de résolution des problèmes. Dans la pratique, vous pouvez commencer à partir d’un modèle sémantique tel que le jeu de données complet Synthea, qui a beaucoup plus de tables. Explorez le jeu de données complet Synthea comme suit.
Lisez tous les fichiers du répertoire synthea/csv :
all_tables = { "allergies": pd.read_csv('synthea/csv/allergies.csv'), "careplans": pd.read_csv('synthea/csv/careplans.csv'), "conditions": pd.read_csv('synthea/csv/conditions.csv'), "devices": pd.read_csv('synthea/csv/devices.csv'), "encounters": pd.read_csv('synthea/csv/encounters.csv'), "imaging_studies": pd.read_csv('synthea/csv/imaging_studies.csv'), "immunizations": pd.read_csv('synthea/csv/immunizations.csv'), "medications": pd.read_csv('synthea/csv/medications.csv'), "observations": pd.read_csv('synthea/csv/observations.csv'), "organizations": pd.read_csv('synthea/csv/organizations.csv'), "patients": pd.read_csv('synthea/csv/patients.csv'), "payer_transitions": pd.read_csv('synthea/csv/payer_transitions.csv'), "payers": pd.read_csv('synthea/csv/payers.csv'), "procedures": pd.read_csv('synthea/csv/procedures.csv'), "providers": pd.read_csv('synthea/csv/providers.csv'), "supplies": pd.read_csv('synthea/csv/supplies.csv'), }Recherchez des relations entre les tables à l’aide de la fonction
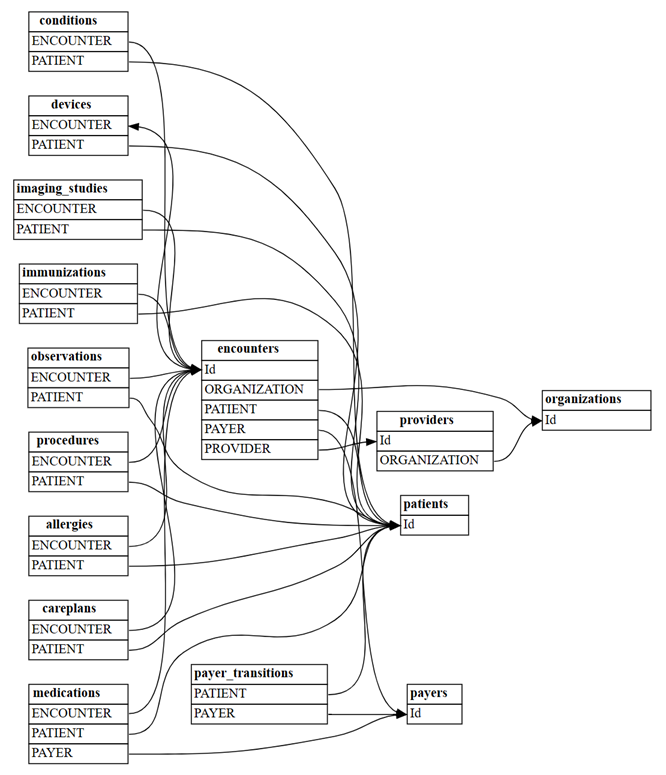
find_relationshipsde SemPy :suggested_relationships = find_relationships(all_tables) suggested_relationshipsVisualisez les relations :
plot_relationship_metadata(suggested_relationships)Comptez le nombre de nouvelles relations « m:m » découvertes avec
include_many_to_many=True. Ces relations sont en plus des relations « m:1 » précédemment affichées, par conséquent, vous devez filtrer surmultiplicity:suggested_relationships = find_relationships(all_tables, coverage_threshold=1.0, include_many_to_many=True) suggested_relationships[suggested_relationships['Multiplicity']=='m:m']Vous pouvez trier les données de relation par différentes colonnes pour mieux comprendre leur nature. Par exemple, vous pouvez choisir de classer la sortie par
Row Count FrometRow Count To, ce qui permet d’identifier les tables les plus volumineuses.suggested_relationships.sort_values(['Row Count From', 'Row Count To'], ascending=False)Dans un autre modèle sémantique, il serait peut-être important de se concentrer sur le nombre de valeurs null
Null Count FromouCoverage To.Cette analyse peut vous aider à comprendre si l’une des relations peut être non valide et si vous devez les supprimer de la liste des candidats.
Contenu associé
Découvrez d’autres tutoriels pour le lien sémantique / SemPy :
- Tutoriel : Nettoyer les données avec des dépendances fonctionnelles
- Tutoriel : Analyser les dépendances fonctionnelles dans un modèle sémantique d’échantillon
- Tutoriel : Découvrir des relations dans le modèle sémantique, à l’aide du lien sémantique
- Tutoriel : Extraire et calculer des mesures Power BI à partir d’un notebook Jupyter