dbscan_dynamic_fl()
Die Funktion dbscan_dynamic_fl() ist eine UDF (benutzerdefinierte Funktion), die ein Dataset mithilfe des DBSCAN-Algorithmus gruppiert. Diese Funktion ähnelt dbscan_fl() nur den Features, die von einer einzelnen numerischen Matrixspalte und nicht von mehreren Skalarspalten bereitgestellt werden.
Voraussetzungen
- Das Python-Plug-In muss im Cluster aktiviert sein. Dies ist für die inline Python erforderlich, die in der Funktion verwendet wird.
- Das Python-Plug-In muss in der Datenbank aktiviert sein. Dies ist für die inline Python erforderlich, die in der Funktion verwendet wird.
Syntax
T | invoke dbscan_fl(, features_col cluster_col, epsilon , min_samples, Metrik metric_params, )
Erfahren Sie mehr über Syntaxkonventionen.
Parameter
| Name | Type | Erforderlich | Beschreibung |
|---|---|---|---|
| features_col | string |
✔️ | Der Name der Spalte, die das numerische Array von Features enthält, die für das Clustering verwendet werden sollen. |
| cluster_col | string |
✔️ | Der Name der Spalte zum Speichern der Ausgabecluster-ID für jeden Datensatz. |
| epsilon | real |
✔️ | Der maximale Abstand zwischen zwei Proben, die als Nachbarn betrachtet werden sollen. |
| min_samples | int |
Die Anzahl der Beispiele in einer Nachbarschaft für einen Punkt, der als Kernpunkt betrachtet werden soll. | |
| metric | string |
Die Metrik, die beim Berechnen des Abstands zwischen Punkten verwendet werden soll. | |
| metric_params | dynamic |
Zusätzliche Schlüsselwortargumente für die Metrikfunktion. |
- Ausführliche Beschreibung der Parameter finden Sie in der DBSCAN-Dokumentation
- Eine Liste der Metriken finden Sie unter Entfernungsberechnungen
Funktionsdefinition
Sie können die Funktion definieren, indem Sie den Code entweder als abfragedefinierte Funktion einbetten oder wie folgt als gespeicherte Funktion in Ihrer Datenbank erstellen:
Definieren Sie die Funktion mithilfe der folgenden Let-Anweisung. Es sind keine Berechtigungen erforderlich.
Wichtig
Eine Let-Anweisung kann nicht alleine ausgeführt werden. Auf sie muss eine tabellarische Ausdrucksanweisung folgen. Informationen zum Ausführen eines funktionierenden Beispiels kmeans_fl()finden Sie unter Beispiel.
let dbscan_dynamic_fl=(tbl:(*), features_col:string, cluster_col:string, epsilon:double, min_samples:int=10, metric:string='minkowski', metric_params:dynamic=dynamic({'p': 2}))
{
let kwargs = bag_pack('features_col', features_col, 'cluster_col', cluster_col, 'epsilon', epsilon, 'min_samples', min_samples,
'metric', metric, 'metric_params', metric_params);
let code = ```if 1:
from sklearn.cluster import DBSCAN
from sklearn.preprocessing import StandardScaler
features_col = kargs["features_col"]
cluster_col = kargs["cluster_col"]
epsilon = kargs["epsilon"]
min_samples = kargs["min_samples"]
metric = kargs["metric"]
metric_params = kargs["metric_params"]
df1 = df[features_col].apply(np.array)
mat = np.vstack(df1.values)
# Scale the dataframe
scaler = StandardScaler()
mat = scaler.fit_transform(mat)
# see https://docs.scipy.org/doc/scipy/reference/spatial.distance.html for the various distance metrics
dbscan = DBSCAN(eps=epsilon, min_samples=min_samples, metric=metric, metric_params=metric_params) # 'minkowski', 'chebyshev'
labels = dbscan.fit_predict(mat)
result = df
result[cluster_col] = labels
```;
tbl
| evaluate python(typeof(*),code, kwargs)
};
// Write your query to use the function here.
Beispiel
Im folgenden Beispiel wird der Aufrufoperator verwendet, um die Funktion auszuführen.
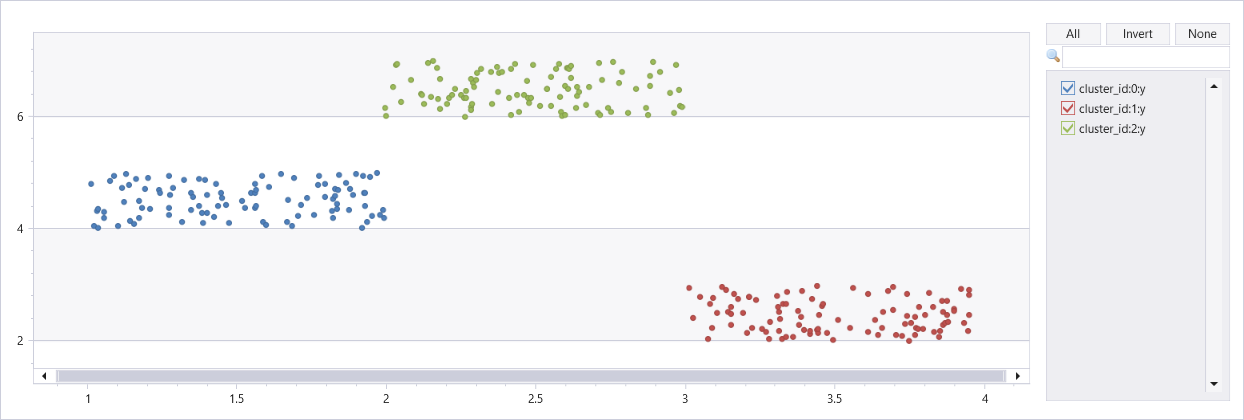
Clustering von künstlichem Dataset mit drei Clustern
Um eine abfragedefinierte Funktion zu verwenden, rufen Sie sie nach der definition der eingebetteten Funktion auf.
let dbscan_dynamic_fl=(tbl:(*), features_col:string, cluster_col:string, epsilon:double, min_samples:int=10, metric:string='minkowski', metric_params:dynamic=dynamic({'p': 2}))
{
let kwargs = bag_pack('features_col', features_col, 'cluster_col', cluster_col, 'epsilon', epsilon, 'min_samples', min_samples,
'metric', metric, 'metric_params', metric_params);
let code = ```if 1:
from sklearn.cluster import DBSCAN
from sklearn.preprocessing import StandardScaler
features_col = kargs["features_col"]
cluster_col = kargs["cluster_col"]
epsilon = kargs["epsilon"]
min_samples = kargs["min_samples"]
metric = kargs["metric"]
metric_params = kargs["metric_params"]
df1 = df[features_col].apply(np.array)
mat = np.vstack(df1.values)
# Scale the dataframe
scaler = StandardScaler()
mat = scaler.fit_transform(mat)
# see https://docs.scipy.org/doc/scipy/reference/spatial.distance.html for the various distance metrics
dbscan = DBSCAN(eps=epsilon, min_samples=min_samples, metric=metric, metric_params=metric_params) # 'minkowski', 'chebyshev'
labels = dbscan.fit_predict(mat)
result = df
result[cluster_col] = labels
```;
tbl
| evaluate python(typeof(*),code, kwargs)
};
union
(range x from 1 to 100 step 1 | extend x=rand()+3, y=rand()+2),
(range x from 101 to 200 step 1 | extend x=rand()+1, y=rand()+4),
(range x from 201 to 300 step 1 | extend x=rand()+2, y=rand()+6)
| project Features=pack_array(x, y), cluster_id=int(null)
| invoke dbscan_dynamic_fl("Features", "cluster_id", epsilon=0.6, min_samples=4, metric_params=dynamic({'p':2}))
| extend x=toreal(Features[0]), y=toreal(Features[1])
| render scatterchart with(series=cluster_id)