series_mv_if_anomalies_fl()
Aplica-se a: ✅Microsoft Fabric✅Azure Data Explorer
A função series_mv_if_anomalies_fl() é uma UDF (função definida pelo usuário) que detecta anomalias multivariadas em série aplicando o modelo de floresta de isolamento do scikit-learn. A função aceita um conjunto de séries como matrizes dinâmicas numéricas, os nomes das colunas de recursos e a porcentagem esperada de anomalias de toda a série. A função cria um conjunto de árvores de isolamento para cada série e marca os pontos que são rapidamente isolados como anomalias.
Pré-requisitos
- O plug-in Python deve ser habilitado no cluster. Isso é necessário para o Python embutido usado na função.
- O plug-in Python deve estar habilitado no banco de dados. Isso é necessário para o Python embutido usado na função.
Sintaxe
T | invoke series_mv_if_anomalies_fl(features_cols anomaly_col [ , score_col [, anomalies_pct [, num_trees [ , samples_pct ]]]], )
Saiba mais sobre as convenções de sintaxe.
Parâmetros
| Nome | Digitar | Obrigatória | Descrição |
|---|---|---|---|
| features_cols | dynamic |
✔️ | Uma matriz que contém os nomes das colunas usadas para o modelo de detecção de anomalias multivariadas. |
| anomaly_col | string |
✔️ | O nome da coluna para armazenar as anomalias detectadas. |
| score_col | string |
O nome da coluna para armazenar as pontuações das anomalias. | |
| anomalies_pct | real |
Um número real no intervalo [0-50] especificando a porcentagem esperada de anomalias nos dados. Valor padrão: 4%. | |
| num_trees | int |
O número de árvores de isolamento a serem criadas para cada série temporal. Valor padrão: 100. | |
| samples_pct | real |
Um número real no intervalo [0-100] especificando a porcentagem de amostras usadas para construir cada árvore. Valor padrão: 100%, ou seja, use a série completa. |
Definição de função
Você pode definir a função inserindo seu código como uma função definida por consulta ou criando-a como uma função armazenada em seu banco de dados, da seguinte maneira:
Defina a função usando a instrução let a seguir. Nenhuma permissão é necessária.
Importante
Uma instrução let não pode ser executada sozinha. Ele deve ser seguido por uma instrução de expressão tabular. Para executar um exemplo funcional de series_mv_if_anomalies_fl(), consulte Exemplo.
// Define function
let series_mv_if_anomalies_fl=(tbl:(*), features_cols:dynamic, anomaly_col:string, score_col:string='', anomalies_pct:real=4.0, num_trees:int=100, samples_pct:real=100.0)
{
let kwargs = bag_pack('features_cols', features_cols, 'anomaly_col', anomaly_col, 'score_col', score_col, 'anomalies_pct', anomalies_pct, 'num_trees', num_trees, 'samples_pct', samples_pct);
let code = ```if 1:
from sklearn.ensemble import IsolationForest
features_cols = kargs['features_cols']
anomaly_col = kargs['anomaly_col']
score_col = kargs['score_col']
anomalies_pct = kargs['anomalies_pct']
num_trees = kargs['num_trees']
samples_pct = kargs['samples_pct']
dff = df[features_cols]
iforest = IsolationForest(contamination=anomalies_pct/100.0, random_state=0, n_estimators=num_trees, max_samples=samples_pct/100.0)
for i in range(len(dff)):
dffi = dff.iloc[[i], :]
dffe = dffi.explode(features_cols)
iforest.fit(dffe)
df.loc[i, anomaly_col] = (iforest.predict(dffe) < 0).astype(int).tolist()
if score_col != '':
df.loc[i, score_col] = iforest.decision_function(dffe).tolist()
result = df
```;
tbl
| evaluate hint.distribution=per_node python(typeof(*), code, kwargs)
};
// Write your query to use the function here.
Exemplo
O exemplo a seguir usa o operador invoke para executar a função.
Para usar uma função definida por consulta, invoque-a após a definição da função inserida.
// Define function
let series_mv_if_anomalies_fl=(tbl:(*), features_cols:dynamic, anomaly_col:string, score_col:string='', anomalies_pct:real=4.0, num_trees:int=100, samples_pct:real=100.0)
{
let kwargs = bag_pack('features_cols', features_cols, 'anomaly_col', anomaly_col, 'score_col', score_col, 'anomalies_pct', anomalies_pct, 'num_trees', num_trees, 'samples_pct', samples_pct);
let code = ```if 1:
from sklearn.ensemble import IsolationForest
features_cols = kargs['features_cols']
anomaly_col = kargs['anomaly_col']
score_col = kargs['score_col']
anomalies_pct = kargs['anomalies_pct']
num_trees = kargs['num_trees']
samples_pct = kargs['samples_pct']
dff = df[features_cols]
iforest = IsolationForest(contamination=anomalies_pct/100.0, random_state=0, n_estimators=num_trees, max_samples=samples_pct/100.0)
for i in range(len(dff)):
dffi = dff.iloc[[i], :]
dffe = dffi.explode(features_cols)
iforest.fit(dffe)
df.loc[i, anomaly_col] = (iforest.predict(dffe) < 0).astype(int).tolist()
if score_col != '':
df.loc[i, score_col] = iforest.decision_function(dffe).tolist()
result = df
```;
tbl
| evaluate hint.distribution=per_node python(typeof(*), code, kwargs)
};
// Usage
normal_2d_with_anomalies
| extend anomalies=dynamic(null), scores=dynamic(null)
| invoke series_mv_if_anomalies_fl(pack_array('x', 'y'), 'anomalies', 'scores', anomalies_pct=8, num_trees=1000)
| extend anomalies=series_multiply(40, anomalies)
| render timechart
Saída
A tabela normal_2d_with_anomalies contém um conjunto de 3 séries temporais. Cada série temporal tem distribuição normal bidimensional com anomalias diárias adicionadas à meia-noite, 8h e 16h, respectivamente. Você pode criar esse conjunto de dados de exemplo usando uma consulta de exemplo.
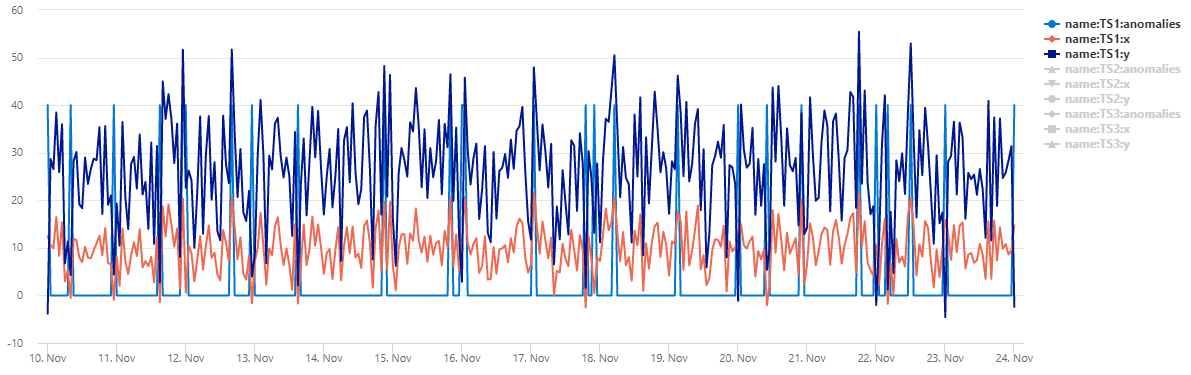
Para exibir os dados como um gráfico de dispersão, substitua o código de uso pelo seguinte:
normal_2d_with_anomalies
| extend anomalies=dynamic(null)
| invoke series_mv_if_anomalies_fl(pack_array('x', 'y'), 'anomalies')
| where name == 'TS1'
| project x, y, anomalies
| mv-expand x to typeof(real), y to typeof(real), anomalies to typeof(string)
| render scatterchart with(series=anomalies)
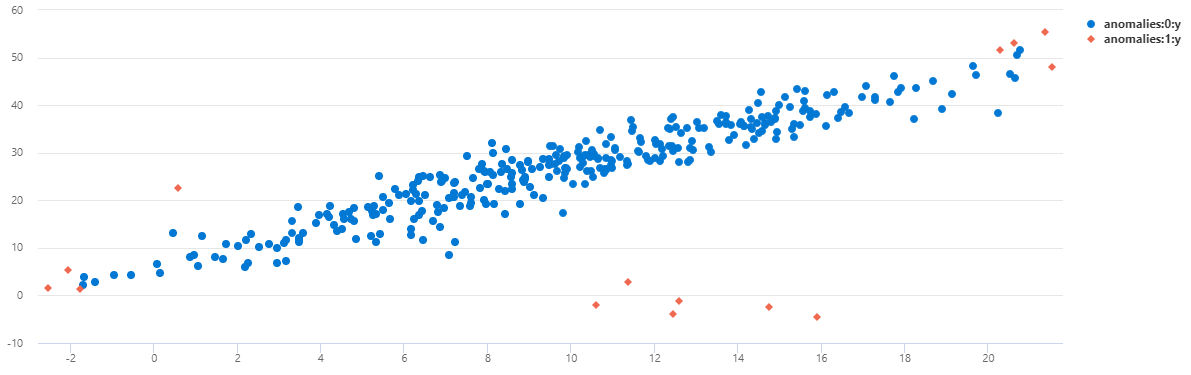
Você pode ver que no TS2 a maioria das anomalias que ocorrem às 8h foram detectadas usando este modelo multivariado.