Visualizações herdadas
Este artigo descreve visualizações herdadas do Azure Databricks. Consulte Visualizações em blocos de anotações Databricks para obter suporte à visualização atual.
O Azure Databricks também suporta, de forma nativa, bibliotecas de visualizações em Python e R e permite instalar e utilizar bibliotecas de terceiros.
Criar uma visualização herdada
Para criar uma visualização herdada a partir de uma célula de resultados, clique em + e selecione Visualização Legada.
As visualizações herdadas suportam um rico conjunto de tipos de plotagem:
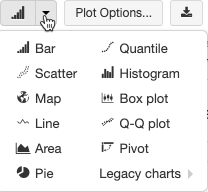
Escolher e configurar um tipo de gráfico herdado
Para escolher um gráfico de barras, clique no ícone  do gráfico de barras :
do gráfico de barras :
Para escolher outro tipo de gráfico, clique ![]() à direita do gráfico
à direita do gráfico  de barras e escolha o tipo de gráfico.
de barras e escolha o tipo de gráfico.
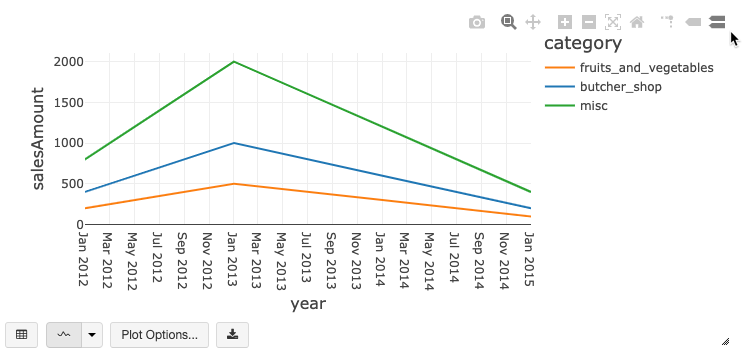
Barra de ferramentas de gráfico herdado
Os gráficos de linhas e de barras têm uma barra de ferramentas interna que suporta um rico conjunto de interações do lado do cliente.
Para configurar um gráfico, clique em Opções de Representação....
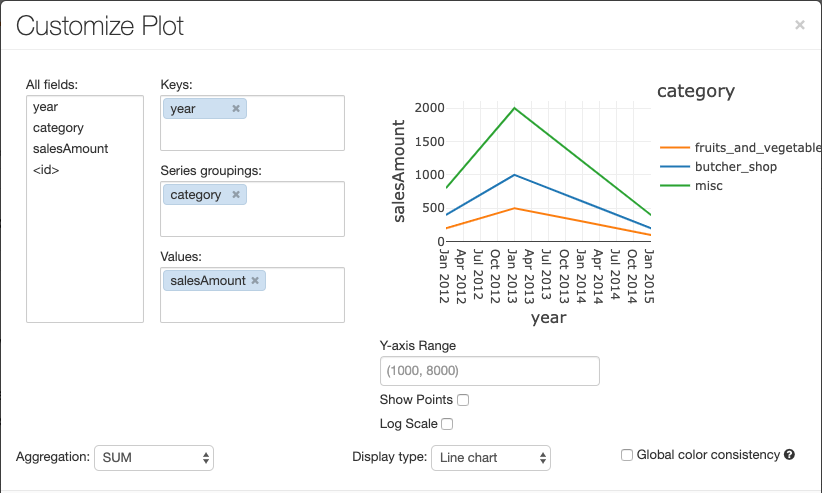
O gráfico de linhas tem algumas opções personalizáveis, como definir um intervalo de eixo Y, mostrar e ocultar pontos e apresentar o eixo Y juntamente com uma escala de registo.
Para obter informações sobre os tipos de gráficos legados, veja:
Consistência de cores transversal aos gráficos
O Azure Databricks dá suporte a dois tipos de consistência de cores em gráficos herdados: conjunto de séries e global.
conjunto de séries consistência de cores atribui a mesma cor ao mesmo valor se você tiver séries com os mesmos valores, mas em ordens diferentes (por exemplo, A = ["Apple", "Orange", "Banana"] e B = ["Orange", "Banana", "Apple"]). Os valores são classificados antes da plotagem, de modo que ambas as legendas são classificadas da mesma maneira (["Apple", "Banana", "Orange"]), e os mesmos valores recebem as mesmas cores. No entanto, se tiveres uma série C = ["Orange", "Banana"], ela não seria consistente em termos de cor com o conjunto A porque o conjunto não é o mesmo. O algoritmo de classificação atribuiria a primeira cor a "Banana" no conjunto C, mas a segunda cor a "Banana" no conjunto A. Se desejar que essas séries sejam consistentes em cores, você pode especificar que os gráficos devem ter consistência de cores global.
Na consistência global de cores , cada valor é sempre associado à mesma cor, independentemente dos valores da série. Para ativar isto em cada gráfico, selecione a caixa de seleção Consistência de cor global.
Nota
Para obter essa consistência, o Azure Databricks hashes diretamente de valores para cores. Para evitar colisões (em que dois valores vão exatamente para a mesma cor), o hash é feito sobre um vasto conjunto de cores, o que resulta no efeito secundário de que não se pode garantir cores bonitas ou facilmente distinguíveis. Com tantas cores, certamente haverá algumas que são muito parecidas.
Visualizações de aprendizado de máquina
Além dos tipos de gráficos padrão, as visualizações herdadas suportam os seguintes parâmetros e resultados de treinamento de aprendizado de máquina:
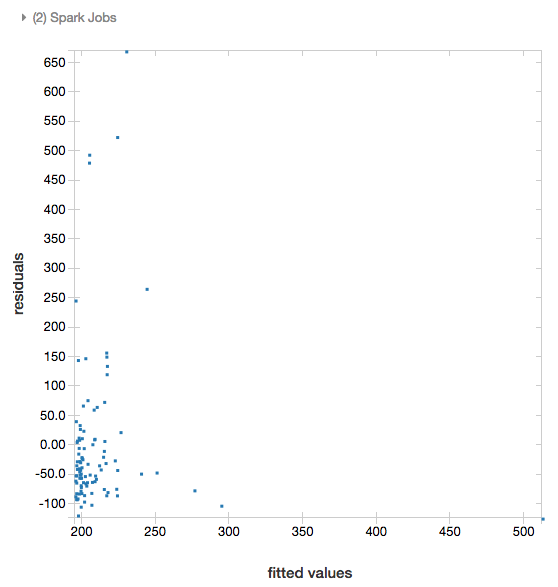
Residuais
Para regressões lineares e logísticas, você pode renderizar um gráfico ajustado versus residual. Para obter este gráfico, forneça o modelo e o DataFrame.
O exemplo seguinte executa uma regressão linear na população da cidade para armazenar dados de preços de venda e, depois, apresenta os valores residuais face aos dados ajustados.
# Load data
pop_df = spark.read.csv("/databricks-datasets/samples/population-vs-price/data_geo.csv", header="true", inferSchema="true")
# Drop rows with missing values and rename the feature and label columns, replacing spaces with _
from pyspark.sql.functions import col
pop_df = pop_df.dropna() # drop rows with missing values
exprs = [col(column).alias(column.replace(' ', '_')) for column in pop_df.columns]
# Register a UDF to convert the feature (2014_Population_estimate) column vector to a VectorUDT type and apply it to the column.
from pyspark.ml.linalg import Vectors, VectorUDT
spark.udf.register("oneElementVec", lambda d: Vectors.dense([d]), returnType=VectorUDT())
tdata = pop_df.select(*exprs).selectExpr("oneElementVec(2014_Population_estimate) as features", "2015_median_sales_price as label")
# Run a linear regression
from pyspark.ml.regression import LinearRegression
lr = LinearRegression()
modelA = lr.fit(tdata, {lr.regParam:0.0})
# Plot residuals versus fitted data
display(modelA, tdata)
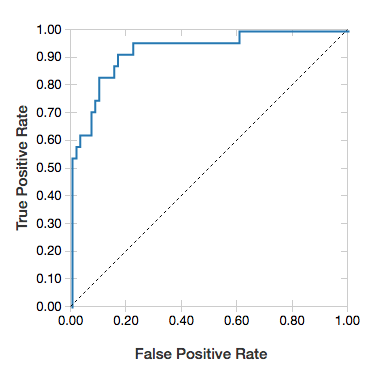
Curvas ROC
Para regressões logísticas, você pode renderizar uma curva ROC . Para obter esse gráfico, forneça o modelo, os dados preparados que são inseridos no fit método e o parâmetro "ROC".
O exemplo a seguir desenvolve um classificador que prevê se um indivíduo ganha <=50K ou >50k por ano a partir de vários atributos do indivíduo. O conjunto de dados Adultos é derivado de dados de recenseamento e consiste em informações de cerca de 48842 pessoas e respetivos rendimentos anuais.
O código de exemplo nesta secção utiliza a codificação "one-hot".
# This code uses one-hot encoding to convert all categorical variables into binary vectors.
schema = """`age` DOUBLE,
`workclass` STRING,
`fnlwgt` DOUBLE,
`education` STRING,
`education_num` DOUBLE,
`marital_status` STRING,
`occupation` STRING,
`relationship` STRING,
`race` STRING,
`sex` STRING,
`capital_gain` DOUBLE,
`capital_loss` DOUBLE,
`hours_per_week` DOUBLE,
`native_country` STRING,
`income` STRING"""
dataset = spark.read.csv("/databricks-datasets/adult/adult.data", schema=schema)
from pyspark.ml import Pipeline
from pyspark.ml.feature import OneHotEncoder, StringIndexer, VectorAssembler
categoricalColumns = ["workclass", "education", "marital_status", "occupation", "relationship", "race", "sex", "native_country"]
stages = [] # stages in the Pipeline
for categoricalCol in categoricalColumns:
# Category indexing with StringIndexer
stringIndexer = StringIndexer(inputCol=categoricalCol, outputCol=categoricalCol + "Index")
# Use OneHotEncoder to convert categorical variables into binary SparseVectors
encoder = OneHotEncoder(inputCols=[stringIndexer.getOutputCol()], outputCols=[categoricalCol + "classVec"])
# Add stages. These are not run here, but will run all at once later on.
stages += [stringIndexer, encoder]
# Convert label into label indices using the StringIndexer
label_stringIdx = StringIndexer(inputCol="income", outputCol="label")
stages += [label_stringIdx]
# Transform all features into a vector using VectorAssembler
numericCols = ["age", "fnlwgt", "education_num", "capital_gain", "capital_loss", "hours_per_week"]
assemblerInputs = [c + "classVec" for c in categoricalColumns] + numericCols
assembler = VectorAssembler(inputCols=assemblerInputs, outputCol="features")
stages += [assembler]
# Run the stages as a Pipeline. This puts the data through all of the feature transformations in a single call.
partialPipeline = Pipeline().setStages(stages)
pipelineModel = partialPipeline.fit(dataset)
preppedDataDF = pipelineModel.transform(dataset)
# Fit logistic regression model
from pyspark.ml.classification import LogisticRegression
lrModel = LogisticRegression().fit(preppedDataDF)
# ROC for data
display(lrModel, preppedDataDF, "ROC")
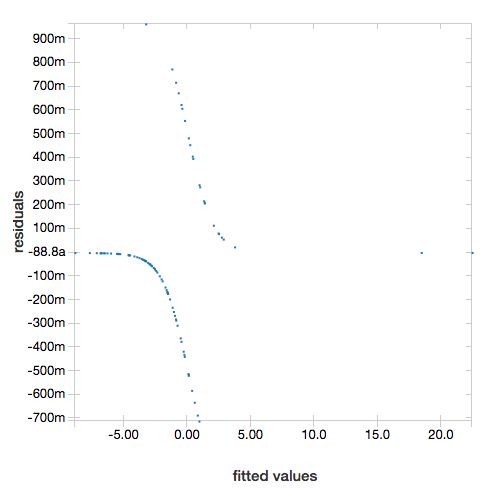
Para apresentar os valores residuais, omita o parâmetro "ROC":
display(lrModel, preppedDataDF)
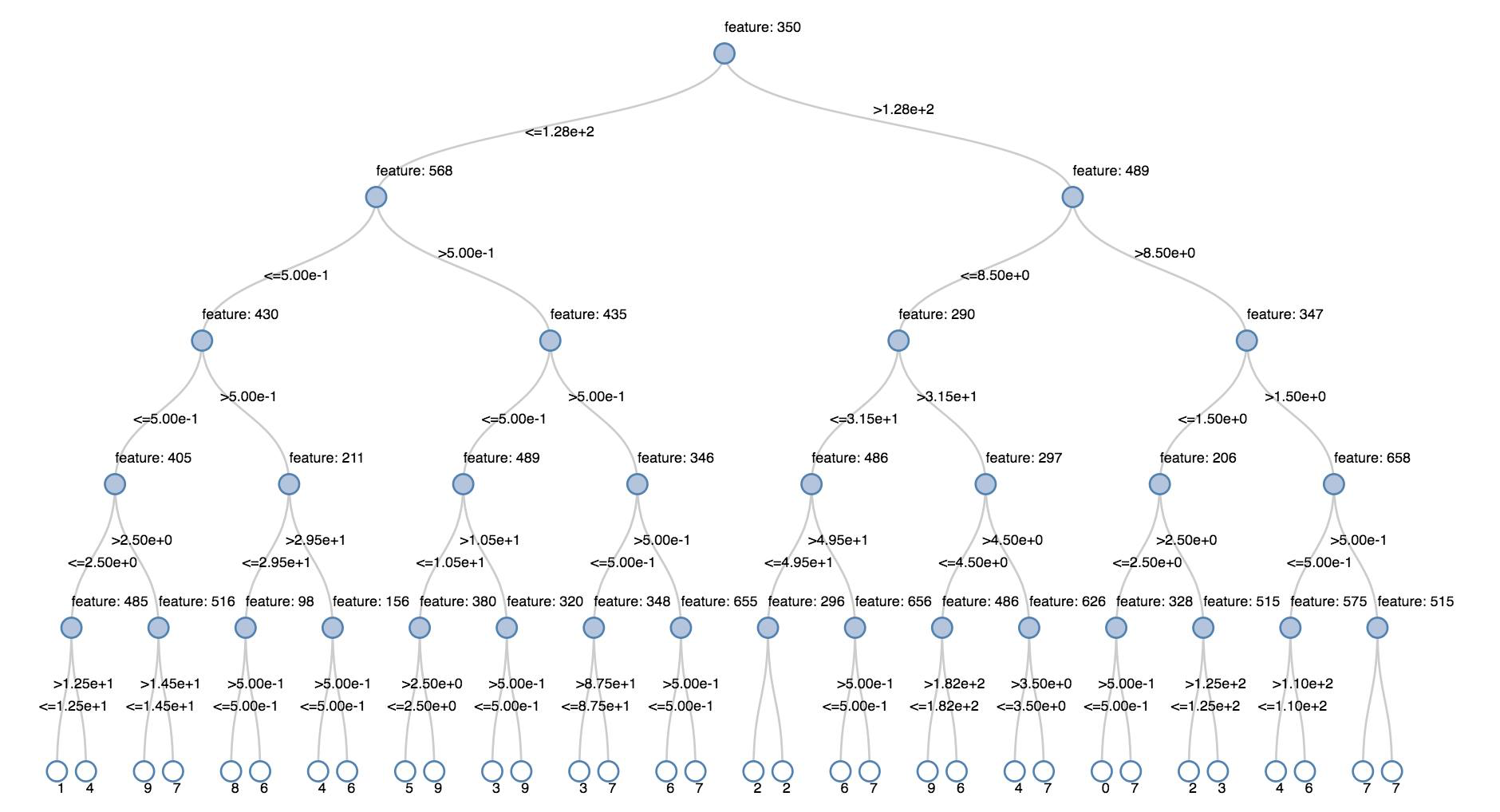
Árvores de decisões
As visualizações herdadas suportam a renderização de uma árvore de decisão.
Para obter essa visualização, forneça o modelo de árvore de decisão.
Os exemplos seguintes preparam uma árvore para reconhecer os dígitos (0 a 9) do conjunto de dados do MNIST de imagens de dígitos manuscritos e, depois, mostra essa árvore.
Python
trainingDF = spark.read.format("libsvm").load("/databricks-datasets/mnist-digits/data-001/mnist-digits-train.txt").cache()
testDF = spark.read.format("libsvm").load("/databricks-datasets/mnist-digits/data-001/mnist-digits-test.txt").cache()
from pyspark.ml.classification import DecisionTreeClassifier
from pyspark.ml.feature import StringIndexer
from pyspark.ml import Pipeline
indexer = StringIndexer().setInputCol("label").setOutputCol("indexedLabel")
dtc = DecisionTreeClassifier().setLabelCol("indexedLabel")
# Chain indexer + dtc together into a single ML Pipeline.
pipeline = Pipeline().setStages([indexer, dtc])
model = pipeline.fit(trainingDF)
display(model.stages[-1])
Scala
val trainingDF = spark.read.format("libsvm").load("/databricks-datasets/mnist-digits/data-001/mnist-digits-train.txt").cache
val testDF = spark.read.format("libsvm").load("/databricks-datasets/mnist-digits/data-001/mnist-digits-test.txt").cache
import org.apache.spark.ml.classification.{DecisionTreeClassifier, DecisionTreeClassificationModel}
import org.apache.spark.ml.feature.StringIndexer
import org.apache.spark.ml.Pipeline
val indexer = new StringIndexer().setInputCol("label").setOutputCol("indexedLabel")
val dtc = new DecisionTreeClassifier().setLabelCol("indexedLabel")
val pipeline = new Pipeline().setStages(Array(indexer, dtc))
val model = pipeline.fit(trainingDF)
val tree = model.stages.last.asInstanceOf[DecisionTreeClassificationModel]
display(tree)
DataFrames de streaming estruturados
Para visualizar o resultado de uma consulta de transmissão em fluxo em tempo real, pode utilizar display para mostrar um DataFrame de Transmissão de Estruturada em Scala e Python.
Python
streaming_df = spark.readStream.format("rate").load()
display(streaming_df.groupBy().count())
Scala
val streaming_df = spark.readStream.format("rate").load()
display(streaming_df.groupBy().count())
display suporta os seguintes parâmetros opcionais:
-
streamName: o nome da consulta de transmissão. -
trigger(Scala) eprocessingTime(Python): define a frequência com que a consulta de transmissão é executada. Se não forem especificados, o sistema verifica a disponibilidade dos dados novos assim que o processamento anterior tiver sido concluído. Para reduzir o custo de produção, o Databricks recomenda que você sempre definir um intervalo de gatilho. O intervalo de gatilho padrão é 500 ms. -
checkpointLocation: o local onde o sistema grava todas as informações do ponto de verificação. Se não for especificado, o sistema gera automaticamente uma localização de ponto de verificação temporária no DBFS. Para que seu fluxo continue processando dados de onde parou, você deve fornecer um local de ponto de verificação. Em produção, o Databricks recomenda que especifique sempre a opçãocheckpointLocation.
Python
streaming_df = spark.readStream.format("rate").load()
display(streaming_df.groupBy().count(), processingTime = "5 seconds", checkpointLocation = "dbfs:/<checkpoint-path>")
Scala
import org.apache.spark.sql.streaming.Trigger
val streaming_df = spark.readStream.format("rate").load()
display(streaming_df.groupBy().count(), trigger = Trigger.ProcessingTime("5 seconds"), checkpointLocation = "dbfs:/<checkpoint-path>")
Para obter mais informações sobre esses parâmetros, consulte Iniciando consultas de streaming.
Função displayHTML
Os blocos de notas de linguagens de programação do Azure Databricks (Python, R e Scala) suportam gráficos HTML mediante a utilização da função displayHTML; pode transmitir à função qualquer código HTML, CSS ou JavaScript. Esta função suporta gráficos interativos que utilizem bibliotecas JavaScript, como D3.
Para obter exemplos da utilização de displayHTML, veja:
Nota
O iframe displayHTML é servido a partir do domínio databricksusercontent.com e o sandbox de iframe inclui o atributo allow-same-origin.
databricksusercontent.com tem de ser acessível a partir do browser. Se ele estiver bloqueado pela sua rede corporativa, ele deverá ser adicionado a uma lista de permissões.
Imagens
As colunas que contêm tipos de dados de imagem são renderizadas como HTML avançado. O Azure Databricks tenta renderizar miniaturas de imagem para DataFrame colunas correspondentes ao Spark ImageSchema.
A renderização de miniaturas funciona para qualquer imagem lida com êxito através da spark.read.format('image') função. Para valores de imagem gerados por outros meios, o Azure Databricks dá suporte à renderização de imagens de 1, 3 ou 4 canais (onde cada canal consiste em um único byte), com as seguintes restrições:
-
Imagens de um canal: o campo
modetem de ser igual a 0. Os camposheight,widthenChannelstêm de escrever corretamente os dados de imagens binários no campodata. -
Imagens de três canais: o campo
modetem de ser igual a 16. Os camposheight,widthenChannelstêm de escrever corretamente os dados de imagens binários no campodata. O campodatatem de conter os dados de píxeis em segmentos de três bytes, com a ordem no canal de(blue, green, red)para cada píxel. -
Imagens de quatro canais: o campo
modetem de ser igual a 24. Os camposheight,widthenChannelstêm de escrever corretamente os dados de imagens binários no campodata. O campodatatem de conter os dados de píxeis em segmentos de quatro bytes, com a ordem no canal de(blue, green, red, alpha)para cada píxel.
Exemplo
Imagine que tem uma pasta que inclui algumas imagens:
Se você ler as imagens em um DataFrame e, em seguida, exibir o DataFrame, o Azure Databricks renderizará miniaturas das imagens:
image_df = spark.read.format("image").load(sample_img_dir)
display(image_df)

Visualizações em Python
Nesta secção:
Seaborn
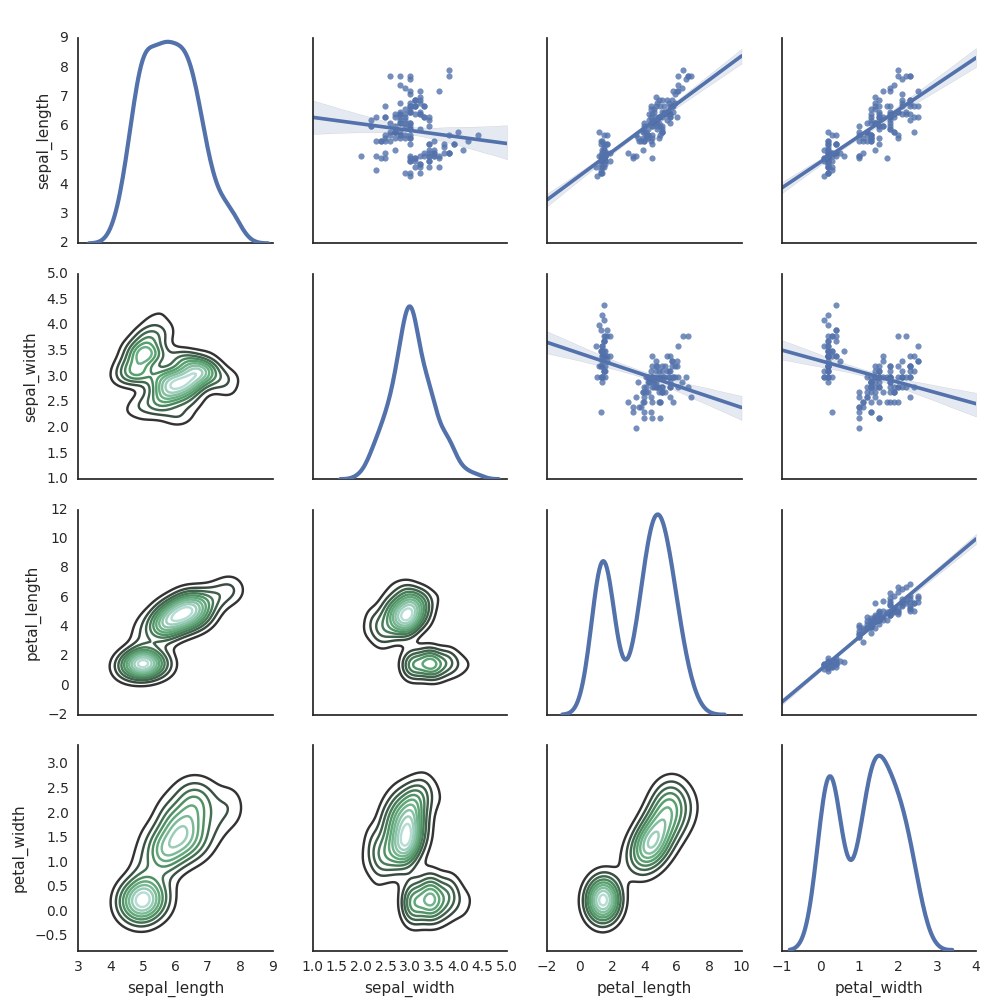
Você também pode usar outras bibliotecas Python para gerar plots. O Databricks Runtime inclui a biblioteca de visualizações Seaborn. Para criar uma representação do Seaborn, importe a biblioteca, crie uma representação e transmita-a à função display.
import seaborn as sns
sns.set(style="white")
df = sns.load_dataset("iris")
g = sns.PairGrid(df, diag_sharey=False)
g.map_lower(sns.kdeplot)
g.map_diag(sns.kdeplot, lw=3)
g.map_upper(sns.regplot)
display(g.fig)
Outras bibliotecas de Python
Visualizações em R
Para representar dados em R, utilize a função display da seguinte forma:
library(SparkR)
diamonds_df <- read.df("/databricks-datasets/Rdatasets/data-001/csv/ggplot2/diamonds.csv", source = "csv", header="true", inferSchema = "true")
display(arrange(agg(groupBy(diamonds_df, "color"), "price" = "avg"), "color"))
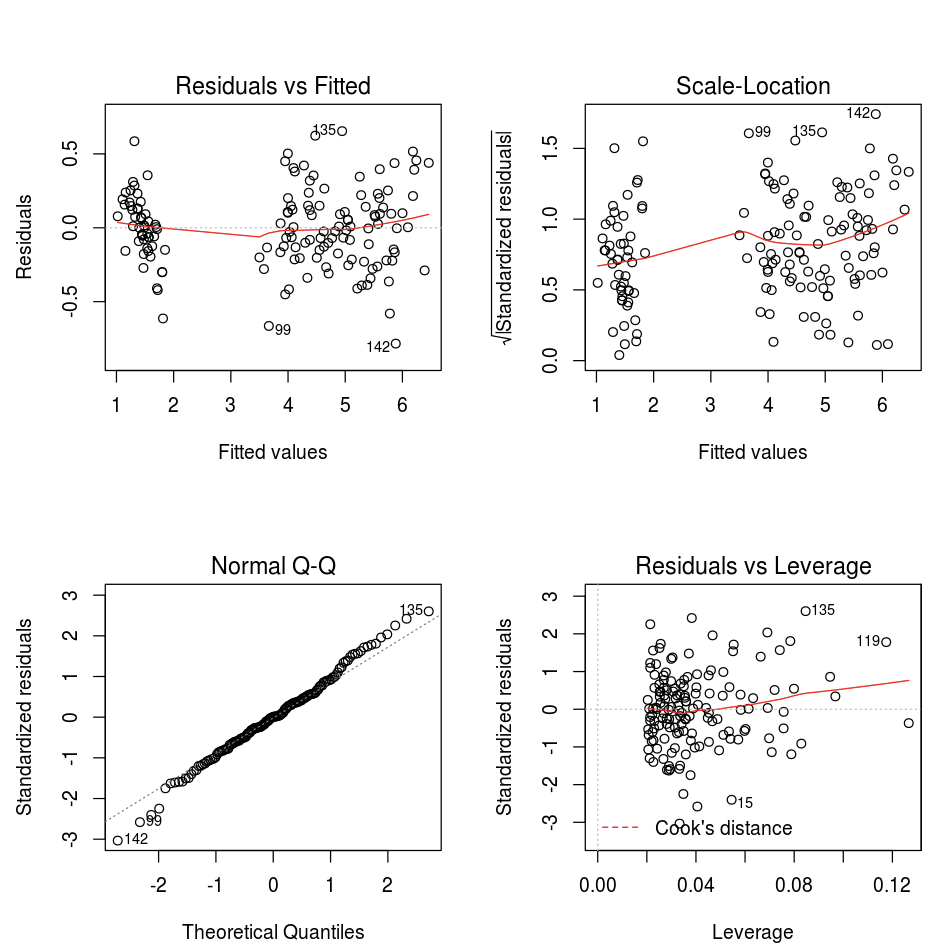
Pode utilizar a função plot predefinida de R.
fit <- lm(Petal.Length ~., data = iris)
layout(matrix(c(1,2,3,4),2,2)) # optional 4 graphs/page
plot(fit)
Também pode utilizar qualquer pacote de visualizações de R. O bloco de notas de R captura a representação resultante como .png e apresenta-o inline.
Nesta secção:
Lattice
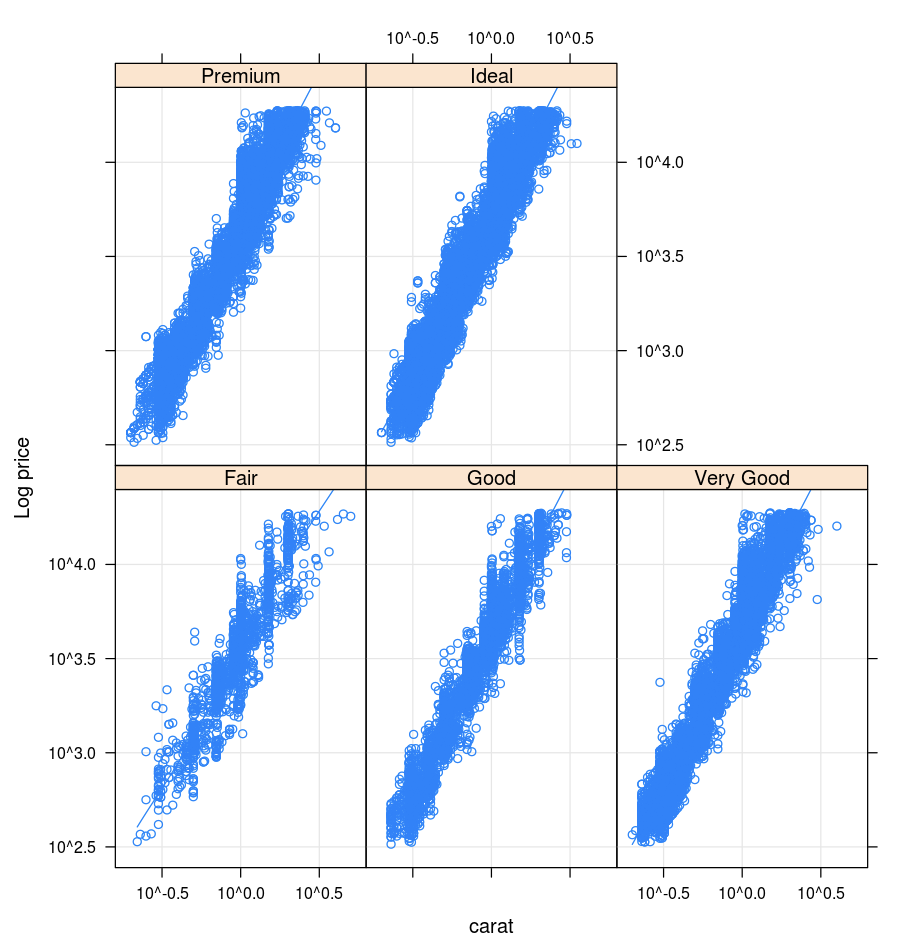
O pacote Lattice suporta gráficos trellis, ou seja, gráficos que apresentam uma variável ou uma relação entre variáveis, condicionadas por uma ou mais variáveis.
library(lattice)
xyplot(price ~ carat | cut, diamonds, scales = list(log = TRUE), type = c("p", "g", "smooth"), ylab = "Log price")
DandEFA
O pacote DandEFA suporta representações "dente de leão".
install.packages("DandEFA", repos = "https://cran.us.r-project.org")
library(DandEFA)
data(timss2011)
timss2011 <- na.omit(timss2011)
dandpal <- rev(rainbow(100, start = 0, end = 0.2))
facl <- factload(timss2011,nfac=5,method="prax",cormeth="spearman")
dandelion(facl,bound=0,mcex=c(1,1.2),palet=dandpal)
facl <- factload(timss2011,nfac=8,method="mle",cormeth="pearson")
dandelion(facl,bound=0,mcex=c(1,1.2),palet=dandpal)
Plotly
O pacote Plotly R depende de htmlwidgets para R. Para obter instruções de instalação e um bloco de anotações, consulte htmlwidgets.
Outras bibliotecas de R
Visualizações em Scala
Para representar dados em Scala, utilize a função display da seguinte forma:
val diamonds_df = spark.read.format("csv").option("header","true").option("inferSchema","true").load("/databricks-datasets/Rdatasets/data-001/csv/ggplot2/diamonds.csv")
display(diamonds_df.groupBy("color").avg("price").orderBy("color"))
Blocos de anotações de mergulho profundo para Python e Scala
Para um mergulho profundo nas visualizações do Python, consulte o bloco de anotações:
Para um mergulho profundo nas visualizações do Scala, consulte o bloco de anotações: